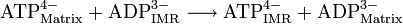- OxPhos
-
 Schematische Darstellung der Elektronentransportkette mit den Komplexen (I, II, III und IV), sowie der ATP-Synthase (Komplex V) in der inneren Membran der Mitochondrien. Oben: Einspeisung der Elektronen über den Komplex I durch Oxidation von NADH zu NAD+. Die Elektronen werden über Coenzym Q, zum Komplex III und weiter über Cytochrom c zum Komplex IV transportiert, wo sie Sauerstoff (O2) zu Wasser reduzieren. Unten: Einspeisung der Elektronen über den Komplex II durch Oxidation von Succinat zu Fumarat. Auch hier werden die Elektronen über Coenzym Q, zum Komplex III und weiter über Cytochrom c zum Komplex IV transportiert, wo sie Sauerstoff (O2) zu Wasser reduzieren.
Schematische Darstellung der Elektronentransportkette mit den Komplexen (I, II, III und IV), sowie der ATP-Synthase (Komplex V) in der inneren Membran der Mitochondrien. Oben: Einspeisung der Elektronen über den Komplex I durch Oxidation von NADH zu NAD+. Die Elektronen werden über Coenzym Q, zum Komplex III und weiter über Cytochrom c zum Komplex IV transportiert, wo sie Sauerstoff (O2) zu Wasser reduzieren. Unten: Einspeisung der Elektronen über den Komplex II durch Oxidation von Succinat zu Fumarat. Auch hier werden die Elektronen über Coenzym Q, zum Komplex III und weiter über Cytochrom c zum Komplex IV transportiert, wo sie Sauerstoff (O2) zu Wasser reduzieren.Die Atmungskette, auch Elektronentransportkette, ist ein wichtiger Teil des Energiestoffwechsels der meisten Lebewesen und besteht aus einer Kette von nacheinander stattfindenden biochemischen Redoxreaktionen. Sie wird auch Atmungskettenphosphorylierung oder oxidative Phosphorylierung (Abkürzung: Oxphos) genannt. Sie läuft sowohl bei Prokaryoten als auch in den Zellen höher entwickelter Lebewesen (Eukaryoten) ab. Bei Eukaryoten findet sie in den Mitochondrien in der inneren Mitochondrienmembran, bei Prokaryoten in der Zellmembran statt. Bei Eukaryoten sind an der Reaktion nacheinander die Enzym-Komplexe I bis V und die Wasserstoff- bzw. Elektronenüberträger Ubichinon (Coenzym Q) und Cytochrom c, die in die innere Mitochondrienmembran eingelagert sind, beteiligt.
Netto läuft dabei die exergonische Knallgasreaktion (Oxidation von Wasserstoff zu Wasser) ab. Tatsächlich werden die durch NADH, FMNH2 und FADH2 angelieferten Reduktionsäquivalente mit Hilfe einer Reihe von Redoxvorgängen, die an der inneren Mitochondrienmembran ablaufen, dazu genutzt, aus ADP und Phosphat die universelle „Energiewährung“ der Zelle, ATP, zu synthetisieren. Diese Reduktionsäquivalente NADH sowie FADH2, sozusagen gebundene Elektronen, entstammen dem Citratzyklus, dem Abbau der Fettsäuren und der Glycolyse.
Dabei spielt nach der chemiosmotischen Theorie auch ein Protonengradient und für die Gesamtausbeute der zuerst von Peter Mitchell postulierte Q-Zyklus die entscheidende Rolle.
Die Elektronentransportkette ist eine Reihe hintereinander geschalteter Redox-Moleküle, die in der Lage sind Elektronen aufzunehmen bzw. abzugeben. Über diese Kette werden Elektronen weitergegeben, sie fallen sozusagen in Stufen bergab, wobei die einzelnen Redox-Moleküle ein zunehmend niedriges Energieniveau haben. Die Elektronentransportkette bezeichnet man auch als Redoxsystem.
Inhaltsverzeichnis
Funktionen der Komplexe der Atmungskette
Ein Mitochondrium enthält außer seiner äußeren noch eine innere Membran. Den Raum zwischen diesen beiden Membranen nennt man Intermembranraum (perimitochondrialer Raum).
Vier der fünf Komplexe der Atmungskette durchspannen jeweils die innere Mitochondrienmembran vollständig, Komplex II hingegen „endet blind“. Es wird ein Protonengradient zwischen dem Intermembranraum und dem Inneren (Matrix) des Mitochondriums erzeugt, der dann in Komplex V zur Synthese von ATP genutzt wird.Komplex I
NADH : Ubichinon – Oxidoreduktase oder NADH Dehydrogenase. Dieser sehr große Enzymkomplex (940 kDa) reduziert mittels Reduktionsäquivalenten NADH, vor allem aus dem Citratcyclus, Ubichinon (UQ) zum Ubihydrochinon, auch Ubichinol (UQH2) genannt. Im Enzymkomplex werden flavinhaltige Nukleotide (FMN) sowie Eisen-Schwefel-Zentren als prosthetische Gruppen benötigt. Pro oxidiertem NADH+H+ werden netto vier Protonen in den Intermembranraum transportiert.
Komplex II
Succinat : Ubichinon – Oxidoreduktase oder Succinat Dehydrogenase. Der Komplex II ist das Enzym Succinat-Dehydrogenase aus dem Citratcyclus. Bei der Reaktion im Citratcyclus wird Succinat zu Fumarat oxidiert. FAD liegt als prosthetische Gruppe im Enzym vor. Es überträgt seine Elektronen im Komplex II auf Ubichinon, das zu Ubihydrochinon reduziert wird. Auch Komplex II enthält Eisen-Schwefel-Zentren, wie Komplex I; allerdings werden keine Protonen in den Intermembranraum gepumpt.
Komplex III
Ubihydrochinon : Cytochrom c – Oxidoreduktase oder Cytochrom c Reduktase. Im Komplex III wird Ubichinol (Coenzym Q) oxidiert und Cytochrom c reduziert. Dabei werden zwei Protonen vom Matrixraum in den Intermembranraum transportiert. Der Mechanismus der Kopplung von Elektronentransfer und Protonentransport im Komplex III wird als Q-Zyklus bezeichnet.
Komplex IV
Cytochrom c : O2 – Oxidoreduktase oder Cytochrom c Oxidase. Im Komplex IV wird Cytochrom c oxidiert und Sauerstoff zu Wasser reduziert. Die dabei freigesetzte Energie wird genutzt um Protonen vom Matrixraum in den Intermembranraum zu pumpen.
Am Komplex IV wird Cytochrom c oxidiert und dabei ein Elektron auf den Komplex übertragen. Nach der sukzessiven Übertragung von vier Elektronen (e−) kann ein gebundenes Sauerstoffmolekül zu zwei Wassermolekülen (H2O) reduziert werden. Die dabei benötigten vier Protonen (H+) werden aus der Matrix entzogen. Die bei der Reduktion von Sauerstoff zu Wasser frei werdende Energie wird vom Enzym genutzt um vier Protonen pro Sauerstoffmolekül von der Matrix über die innere Mitochondrienmembran in den Intermembranraum zu pumpen. Die Cytochrom c Oxidase ist ein Transmembranprotein mit zwei Häm a Molekülen (Häm a und Häm a3) als prosthetische Gruppen und zwei Kupfer-Zentren (CuA und CuB) als Kofaktoren. Das Enzym ist für nahezu sämtlichen Sauerstoffverbrauch aller sauerstoffatmenden Organismen verantwortlich.
Komplex V
FoF1-ATP-Synthase. Der Komplex V, der schließlich ATP synthetisiert, besteht aus zwei Teilen: Der F1-Teil ragt in die Matrix des Mitochondriums hinein, während der Fo-Teil einen Protonen-Kanal durch die innere Mitochondrienmembran bildet. Mit Hilfe der vorangeschalteten vier Komplexe der Atmungskette wurde ein elektrochemischer Protonengradient aufgebaut. Nach den Gesetzen der Thermodynamik besteht somit eine Triebkraft der Protonen im Intermembranraum zur Matrix des Mitochondriums hin. Den passenden Kanal stellt die Fo-Untereinheit dar. Vergleichbar mit einer Turbine – und tatsächlich konnte eine Rotation des Fo-Teils nachgewiesen werden – wird durch den Protonenfluss Energie erzeugt, die zur Synthese von ATP durch die F1-Untereinheit verwendet wird. (siehe Mechanismus der ATP-Synthase)
Strittig ist noch die Frage, wie viele Protonen für die Synthese eines ATP Moleküls aus ADP und anorganischem Phosphat benötigt werden. Nach Löffler, Petrides: mindestens drei, wobei bei der Bilanzierung ein weiteres Proton (also insgesamt mindestens vier) auftaucht, das für den Transport von Phosphat (durch Phosphat/H+-Symport) in den Matrixraum benötigt wurde.
Je nachdem, aus wie vielen c-Untereinheiten der Transmembranring (Fo c-Ring)besteht, müssen unterschiedlich viele Protonen durch Fo in die Matrix transportiert werden. Es können 10-14 Untereinheiten sein. F1 bildet pro 120° Drehung der Gamma-Einheit 1 Molekül ATP - also bei einer ganzen Umdrehung (360°) 3 Moleküle ATP. Die Anzahl der benötigten Protonen für die Bildung eines ATP-Moleküls berechnet sich also wie folgt: 10 c-Einheiten / 3 ATP pro Drehung = 3,33 benötigte Protonen, 14 c-Einheiten / 3 ATP pro Drehung = 4,66 benötigte Protonen ....
Hemmstoffe der Atmungskette
- Rotenon hemmt den Komplex I (NADH Reduktase); es liegt nur eine eingeschränkte Hemmung vor, denn Substrate nach dem Komplex I (Succinat im Komplex II), können ihre Elektronen in die Atmungskette schleusen.
- Malonat hemmt den Komplex II (Succinat Dehydrogenase);
- Antimycin A (Antibiotikum) hemmt den Komplex III (Cytochrom c Reduktase); blockiert die Übertragung von Elektronen vom Coenzym Q auf Cytochrom c; die Komponenten der Atmungskette vor dem Wirkungsort von Antimycin A im Komplex III bleiben reduziert, alle dahinter, bleiben oxidiert; der Verbrauch von Sauerstoff im Komplex IV und die Synthese von ATP im Komplex V wird gehemmt.
- Cyanide, Azide und Kohlenmonoxid hemmen den Komplex IV (Cytochrom c Oxidase); blockieren die Bindungsstelle für Sauerstoff; Folge ist ein Elektronenstau, wodurch die Komponenten der Atmungskette vollständig reduziert vorliegen; der Erhalt des Protonengradienten wird unmöglich; eine ATP-Synthese ist nicht möglich.
 Oligomycin A, ein Makrolidantibiotikum, der die ATP-Synthase hemmt.
Oligomycin A, ein Makrolidantibiotikum, der die ATP-Synthase hemmt.- Oligomycin (Antibiotikum) hemmt den Komplex V (ATP-Synthase); dadurch wird der Protonengradient wesentlich langsamer abgebaut; deshalb nimmt der Elektronenfluss zum Erhalt dieses Gradienten deutlich ab und der Sauerstoffverbrauch sinkt.
Alle bisher angesprochenen Hemmstoffe der Atmungskette bringen einen verminderten Sauerstoffverbrauch mit sich. Anders verhalten sich Hemmstoffe, die als Entkoppler bezeichnet werden.
Entkoppler der Atmungskette
Entkoppler (Protonophore) erzwingen einen maximalen Sauerstoffverbrauch, jedoch ohne die Synthese von ATP. Sie bauen das mitochondriale Membranpotential (Protonengradient) ab.
- natürliche Entkoppler:
- Thermogenin. Siehe unter braunes Fettgewebe.
- künstliche Entkoppler:
- 2,4-Dinitrophenol ist eine schwache Säure. Sie wird über der inneren Mitochondrienmembran aufgrund des reichen Vorkommens von H+-Ionen (niedriger pH) protoniert, kann dann die Membran passieren und wird intramitochondrial (höherer pH) deprotoniert. Der Protonengradient wird so abgebaut. Um diesen zu erhalten, arbeiten die Komplexe auf Hochtouren. So kommt es zu einem maximalen Sauerstoffverbrauch.
- CCCP, FCCP
ATP/ADP-Translokase
Das durch Komplex V erzeugte ATP kann die innere Mitochondriummembran nicht frei passieren. Umgekehrt kann das für die ATP-Synthase benötigte ADP nicht aus dem Cytosol in die Matrix diffundieren. Jedoch existiert ein spezielles Transportsystem, um ATP in das Cytosol und ADP in die Matrix zu bringen. Eine 30 kD große ATP/ADP-Translokase, auch Adeninnukleotid-Translokase (ANT), überführt ein Molekül ATP aus der Matrix in den Intermembranraum (IMR) und gleichzeitig umgekehrt ein Molekül ADP aus den IMR in die Matrix. Die Translokase ist damit ein Antiporter und wird dadurch betrieben, dass ADP bzw. ATP ohne bindende Magnesiumionen transportiert werden.[1] ATP ist bei pH 7 damit vierfach negativ geladen, ADP dreifach negativ. Damit wandert ATP bevorzugt entlang des Membranpotentials aus der Matrix in den IMR.[1]
Der Antiporter ist ein häufiges Protein der inneren Membran des Mitochondriums und kann bis zu 14 % aller Proteine der inneren Membran ausmachen.[2] Infolgedessen ist es unwahrscheinlich, dass die ATP-Synthese aus Mangel an ADP limitiert ist. Die Translokase kann durch Atractylosid, einem Glykosid, gehemmt werden. Auch Bongkreksäure inhibiert den Translokator. Diese bindet an der Matrixseite des Proteins und muss infolgedessen erst in die Mitochondriummatrix gelangen. Bongkreksäure wird vom Bakterium Pseudomonas cocovenenas ausgeschieden.[3]
Phosphattransporter
Für die Synthese von ATP aus ADP wird auch Phosphat benötigt. Dieses kann ebenso wenig durch die innere Membran des Mitochondriums diffundieren wie ATP bzw. ADP. Der Transport wird durch einen speziellen Symporter ermöglicht, der gleichzeitig ein Proton und ein Phosphatmolekül aus dem Intermembranraum in die Matrix transportiert. Dessen Aktivität kann durch Mersalyl inhibiert werden.
Trivia
Harold Baum hat ein Textbuch[4] herausgebracht, in dem biochemische Prozesse durch bekannte Lieder vertont wurden. Dies soll es Studenten erleichtern, sich besser an komplizierte Sachverhalte bei Stoffwechselwegen zu erinnern.
Quellen
- ↑ a b Jeremy M. Berg, Lubert Stryer und John L. Tymoczko: Biochemie. Spektrum Akademischer Verlag; 6. Auflage 2007; ISBN 978-3-8274-1800-5; S. 589
- ↑ Thomas M. Devlin (Hrsg.): Textbook of Biochemistry with Clinical Correlations. Wiley & Sons; 6. Auflage 2005; ISBN 0-471-67808-2; S.570
- ↑ Henderson, PJ. und Lardy, HA. (1970): Bongkrekic acid. An inhibitor of the adenine nucleotide translocase of mitochondria. In: J Biol Chem.245(6); 1319–1326; PMID 4245638; PDF (freier Volltextzugriff, engl.)
- ↑ Harold Baum: Biochemists' Song Book. Taylor & Francis Ltd, 2. Auflage 1998; ISBN 0-7484-0416-3
Literatur
- Saraste, M. (1999). Oxidative phosphorylation at the fin de siecle. In: Science. Bd. 283, Nr. 5407, S. 1488-1493. PMID 10066163
- Löffler, Petrides: Biochemie des Menschen, 7. Aufl
Siehe auch
Weblinks
- Atmungskette (leicht verständliche Darstellung der Atmungskette)
- KEGG: Oxidative phosphorylation - Reference pathway (graphische Darstellung der mitochondrialen Atmungsketten-Komplexe, engl.)
- Mitochondrial Pathways: Oxidative Phosphorylation (engl.)
- The chemical logic behind respiration and fermentation (engl.)
- Elektronentransportkette (animierte Darstellung)
Wikimedia Foundation.