- Signaltransduktionskaskade
-
Die visuelle Signaltransduktionskaskade bezeichnet die Umwandlung eines äußeren Lichtreizes in ein physiologisches Signal im Organismus. Sie findet in den Photorezeptorzellen in der Netzhaut des Auges statt. Das hier geschilderte Prinzip bezieht sich auf Wirbeltiere, findet aber in ähnlicher Form auch bei den meisten anderen Tieren statt.
Inhaltsverzeichnis
Aufbau der Rezeptorzellen
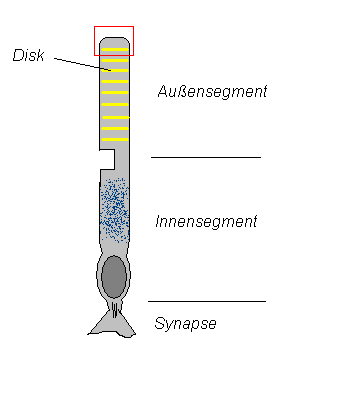
Beteiligt sind hauptsächlich mehrere Proteine im Außensegment der Photorezeptorzellen (Abb.1): Rhodopsin (Eine Verbindung zwischen Opsin und 11-cis-Retinal), Transducin, eine cGMP-Phosphodiesterase sowie Na+/Ca2+-Kanäle in der Photorezeptorzelle. Das Innensegment enthält die Mitochondrien und ist für den Metabolismus der Zelle zuständig.
Das in das Auge einfallende Licht trifft auf das Rhodopsin, das in den Diskmembranen in hoher Konzentration (etwa 30 000 Moleküle/µm²) enthalten ist. Disks sind flache, dichtgepackte Vesikel im Inneren des Außensegments der Rezeptorzelle.
Ablauf der Signaltransduktion
Die Signaltransduktionskaskade
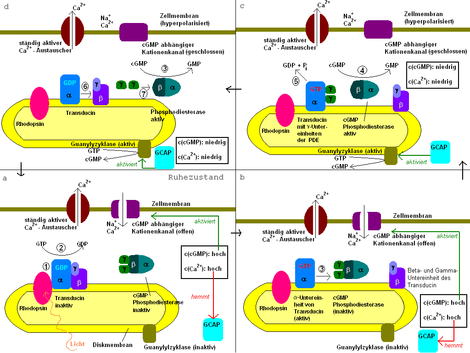
Der genaue Ablauf ist in Abb.2 dargestellt:
(1) Das einfallende Licht wird von 11-cis-Retinal, das über eine Schiff-Base-Bindung im hydrophoben Inneren des Opsin an dieses gebunden ist, absorbiert (Rhodopsin ist eine Verbindung von Opsin und 11-cis-Retinal). Dabei isomerisiert das 11-cis-Retinal zum all-trans-Retinal. Daraufhin wird das Rhodopsin über mehrere Zwischenzustände aktiviert. Das aktivierte Rhodopsin bindet dann die alpha-Untereinheit des Transducins.
(2) Diese Bindung induziert in der α-Untereinheit des Transducins den Austausch von GDP gegen GTP. Dies führt im Weiteren dazu, dass die β/γ-Untereinheit abdissoziiert und die α-Untereinheit aktiv wird.
(3) Die α-Untereinheit des Transducins spaltet die beiden γ-Untereinheiten der cGMP Phosphodiesterase (PDE) ab, bindet sie und aktiviert damit die PDE. Die Abspaltung einer γ-Untereinheit würde zu einer partiellen Aktivierung der PDE führen.
(4) Die aktive PDE spaltet nun cGMP in GMP. Der sinkende cGMP-Spiegel hemmt den Kationen-Einstrom in die Zelle. Die sinkende Ca2+-Konzentration aktiviert nun das Guanylylzyklase-aktivierende Enzym, welches seinerseits die Guanylylzyklase aktiviert. Dadurch wird nun auch cGMP wieder aufgebaut, es stellt sich also ein Gleichgewicht zwischen Auf- und Abbau ein.
(5) Nach einiger Zeit spaltet die intrinsische GTPase der α-Untereinheit das GTP in GDP und Phosphat. Dadurch werden die γ-Untereinheiten der PDE wieder freigegeben.
(6) Die so regenerierte α-Untereinheit lagert sich nun wieder mit der β/γ-Untereinheit zusammen und bildet den ursprünglichen Transducin-Komplex.
(7) Die γ-Untereinheiten binden wieder an die Phosphodiesterase und inaktiviert sie damit. Deshalb wird nun nur noch cGMP aus GTP synthetisiert und die cGMP und damit die Ca2+-Konzentration steigen an (siehe unten).
Regeneration des Systems
Aktiviertes Rhodopsin (auch Metarhodopsin II) zerfällt zwar nach einiger Zeit in seinen Proteinanteil Opsin und all-trans-Retinal. Letzteres wird mit einer Isomerase wieder in 11-cis-Retinal umgewandelt, welches dann erneut an Opsin binden kann. Allerdings dauert dieser Prozess zu lange. Daher wird Rhodopsin über folgende Reaktionsfolge inaktiviert und regeneriert: Rhodopsin wird durch eine spezifische Kinase phosphoryliert. An das phosphorylierte Rhodopsin bindet nun Arrestin. Das Arrestin dissoziiert erst wieder vom Rhodopsin, wenn das all-trans-Retinal durch Retinoldehydrogenase zu all-trans-Retinol reduziert wurde. Anschließend wird das Opsin durch die Phosphatase 2A dephosphoryliert. Das Rhodopsin kann nun mit 11-cis-Retinal regeneriert werden. Durch diese Inaktivierung wird verhindert, dass einmal aktiviertes Rhodopsin die Signalkaskade zu lange aufrecht erhält.
Es findet, wie oben bereits angesprochen, auch ein rückgekoppelter Regelkreislauf über den Ca2+-Spiegel in der Zelle statt (Abb.2, Abb.3), die auch an der Regeneration und der Adaptation dieser Prozesse beteiligt ist. Sind die Ionenkanäle geschlossen, strömt kein Ca2+ mehr in die Zelle und der ständig aktive Ca2+-Austauscher befördert Ca2+ aus der Zelle heraus, so dass die Ca2+-Konzentration sinkt. Dieses bewirkt eine Steigerung der Aktivität des Guanylylzyklase-aktivierenden-Enzyms (GCAP) (auch: Guanylatcyclase-aktivierendes-Enzym), die von Ca2+-Ionen inhibiert wird. GCAP aktiviert nun eine cGMP-synthetisierenden Guanylylzyklase und der niedrige cGMP-Spiegel wird wieder auf altes Niveau gebracht. Na+-Ca2+-Kanäle öffnen sich wieder durch das cGMP und der Ca2+-Spiegel steigt wieder, wodurch die Aktivität von GCAP und gleichzeitig auch die Guanylylzyklase wieder nachlässt usw. Es entsteht also ein cGMP-Gleichgewicht aus dem Abbau durch die cGMP-PDE und die Synthese von cGMP durch die Guanylylzyklase.
Der entstehende Impuls kann hierdurch auch über den Ca2+-Spiegel reguliert werden und trägt so zur Adaptation an Lichtverhältnisse bei (z.B. durch pH-abhängige Ca2+-Kanäle). Ist der Lichtreiz jedoch vorbei, stoppt die Aktivität der PDE relativ schnell durch die Regeneration von Transducin (von Abschnitt d zu Abschnitt a in Abb. 2). Die Guanylylzyklase synthetisiert nun cGMP, so dass dessen Konzentration wieder auf das normale Maß steigt. Dies aktiviert auch wieder den cGMP-abhängigen Kationentransporter und der Dunkelstrom fließt wieder. Auch der Ca2+-Spiegel steigt wieder und stoppt so indirekt die Guanylylzyklase. Das System ist bereit für den nächsten Lichtimpuls.
Signalweiterleitung
Im Dunkeln erfolgt eine fortwährende Ausschüttung des Neurotransmitters Glutamat in den Photorezeptoren. Dieser wirkt erregend auf die Postsynapsen von Horizontal- und Bipolarzellen. Durch die Schließung der Kationenkanäle in der Zellmembran des Photorezeptors und der darauf folgenden Hyperpolarisation wird der Neurotransmitter Glutamat nicht weiter ausgeschüttet. In der Folge werden die hemmenden Ionenkanäle der Horizontal- und Bipolarzellen geschlossen. Dadurch können in den Ganglienzellen wieder Aktionspotentiale entstehen. Dieses ist das eigentliche elektrische Signal, das in der Netzhaut moduliert und schließlich weiter geleitet wird in die visuellen Zentren im Gehirn.
Quellen
http://edoc.hu-berlin.de/dissertationen/biologie/tellgmann-gabriele/HTML/tellgmann-ch1.html
Siehe auch
Wikimedia Foundation.