- Small RNA
-
Kleine RNAs, auch Small RNAs sind kurze Moleküle der Ribonukleinsäure (RNA) mit einer Länge von 21–28 Nukleotiden. Sie kommen in eukaryotischen Zellen neben der RNA mit den Funktionen als mRNA, tRNA, rRNA und snRNA vor. Dabei handelt es sich um eine heterogene Gruppe von RNAs unterschiedlicher Herkunft und Funktion. Aufgrund ihrer Entdeckungsgeschichte tragen diese RNAs auch bei gleicher Herkunft und Funktion je nach Organismus zum Teil unterschiedliche Bezeichnungen.
Kleine RNAs werden aus doppelsträngigen Vorläufern durch Proteine mit RNasedomänen (wie z. B. Dicer und Drosha/DGCR8) herausgeschnitten und erfüllen wichtige Funktionen bei der Regulation von zellulären Prozessen wie etwa Wachstumsregulation, Differenzierung und Apoptose (programmierter Zelltod) oder der Abwehr von Viren.
Bekannte Vertreter sind:
- miRNAs (micro RNAs),
- siRNAs (small interfering RNAs) und
- tnRNAs (tiny noncoding RNAs)
Inhaltsverzeichnis
miRNAs
- Hauptartikel: MicroRNA
microRNA (von griech. micros "klein") sind laut aktueller Definition[1] einzelsträngige RNAs mit etwa 22 Nukleotiden Länge, die von Dicer aus einem Hairpin einer endogenen RNA prozessiert werden. miRNAs beeinflussen die Translation und den Abbau ihrer Ziel-mRNAs, die aufgrund ihrer teilweisen Komplementarität erkannt werden. miRNAs liegen in der Zelle assoziiert mit Proteinen als Ribonucleoproteine (RNP) vor. Die miRNAs erfüllen hier (ähnlich wie z. B. bei den snoRNAs) eine Funktion als guide RNA, d. h. sie „zeigen“ den Proteinen die richtigen Ziel-RNAs. Diese werden dann in ihrer Translation gehemmt oder ähnlich wie bei den siRNAs gespaltet und abgebaut. miRNAs spielen bei der Steuerung einer Vielzahl von zellulären Prozessen eine entscheidende Rolle: bei Pflanzen regulieren sie Wachstum und Blütenbildung, in Drosophila melanogaster den programmierten Zelltod, in menschlichen Zellen die Differenzierung von potentiell unsterblichen Stammzellen zu spezialisierten Geweben und vieles mehr. Auch bei einigen Krebserkrankungen scheinen miRNAs involviert zu sein.
Die Biogenese der miRNAs besteht im Vergleich zu den unten beschriebenen siRNAs aus zwei Prozessierungsschritten. Nach der Transkription des "host"-Gens durch die RNA-Polymerase bildet sich eine "hairpin loop"-Struktur (Haarnadelschleife), die pri-miRNA. Diese wird noch im Zellkern durch den Enzymkomplex Drosha/Pasha (Microprocessor complex) erkannt und prozessiert. Drosha ist wie das Protein Dicer eine Typ III RNase, die den RNA Doppelstrang an festgelegten Positionen spaltet, was zur Freisetzung des jetzt als pre-miRNA bezeichneten hairpins führt. Dieser wird anschließend über einen Exportin 5 und RanGTP vermittelten Transport in das Cytoplasma exportiert, wo er nun als Dicer-Substrat ähnlich den siRNAs zur reifen miRNA geschnitten wird. Zu beachten ist hier, dass genau wie bei den siRNAs bevorzugt ein Strang (die spätere in der Zelle nachweisbare miRNA) in den späteren aktiven Proteinkomplex eingebaut wird. Bei diesem Vorgang spielt das Protein R2D2 eine große Rolle.
Die Zahl der Gene korreliert nur unwesentlich mit der Komplexität eines Lebewesens. Die Fruchtfliege besitzt ca. 13.600 Gene, der Mensch noch nicht einmal doppelt so viele. Hingegen steigt die Gesamtlänge der DNA mit der Entwicklungsstufe. So kodieren nur 2 Prozent der menschlichen DNA Proteine. Die Introns, die mehr als 90 Prozent der DNA ausmachen, hielt man lange Zeit nur für Endmarker zwischen den Genen. Sie scheinen aber Informationen für die mi-RNA zu enthalten, die hauptverantwortlich sind für die Steuerung der Gene. Möglicherweise erhält man aus ihrer Anzahl Rückschlüsse über die Phylogenese.
siRNAs
Small interfering RNAs (siRNAs) sind 21-28 Nukleotide lange RNAs, die von der RNase III Dicer aus langen doppelsträngigen RNAs herausgeschnitten werden. Auch kleine einzelsträngige RNAs, die in der RNA Interferenz (RNAi) benutzt werden, können, auch wenn sie synthetisch hergestellt wurden, als siRNAs bezeichnet werden.
Untergruppen:
- tasiRNA (trans-acting siRNA)
- rasiRNA (repeat-associated siRNA)
- scnRNA (small-scan RNA)
Mechanismus zur siRNA
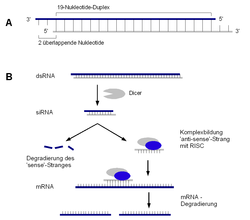
Die siRNAs entstehen durch das Zerschneiden einer längeren dsRNA (doppelsträngigen RNA). Im natürlichen Ablauf vollzieht sich dies im Cytoplasma einer Zelle durch das Enzym Dicer. Die siRNAs können jedoch ebenfalls durch die PEI-Komplexierung direkt von Außen in die Zelle eingebracht werden. Diese kleinen doppelsträngigen RNAs werden anschließend in einen Proteinkomplex RISC (RNA-induced silencing complex) eingebaut. Mithilfe der inkorporierten RNA-Fragmente bindet RISC komplementär an der Ziel-mRNA. So wird der RISC-Komplex mit seinen RNA-Helicase- und Nuklease-Aktivitäten in räumliche Nähe gebracht. Die Folge ist ein Entwinden und Spalten der Ziel-mRNA. Da diese mRNA nun in ungeschützter Form vorliegt, wird sie schnell durch intrazelluläre Nukleasen abgebaut. Somit kann gleichsam Fremd-RNA abgebaut werden, zum Beispiel die von Viren, aber auch der zellinternen Regulation (tasiRNAs: trans-acting siRNAs, rasiRNAs: repeat-associated siRNAs). Die siRNAs werden aktuell intensiv auf ihre Beteiligung an verschiedenen Zellvorgängen (Apoptose, Genregulation, Prionen etc.) und Krankheiten wie Krebs erforscht.
Die molekularbiologische Forschung setzt synthetisch hergestellte siRNAs ein, um durch RNA Interferenz (RNAi) die Expression von spezifischen Zielgenen zu verringern. Die siRNA werden in isolierte Zellen eingebracht (transfiziert) und die mRNA des Zielgens wird abgebaut. Die resultierende Verringerung der Genprodukte (Gene-Knockdown) ermöglicht es, Hinweise auf die physiologische Bedeutung des betreffenden Genes zu erhalten. 2001 wurde diese Technik im Max-Planck-Institut für biophysikalische Chemie in Göttingen in der Arbeitsgruppe um Tom Tuschl erstmals an Säugerzellen erfolgreich durchgeführt.
Einzelnachweise
- ↑ V. Ambros u. a.: A uniform system for microRNA annotation. In: RNA. 9(3) 2003, S. 277-279. PMID 12592000
Siehe auch
Wikimedia Foundation.