- Substitutions Matrix
-
In der Bioinformatik beschreiben die Einträge in einer Substitutionsmatrix eine relative Rate, mit welcher im Laufe der Evolution eine Aminosäure in eine andere mutiert (für den Fall einer Protein-Matrix). Dabei gibt der Eintrag aij die relative Rate an, mit welcher die Aminosäure i zu der Aminosäure j mutiert. Manche Matrizen sind symmetrisch, es gilt also aij = aji. Eine Substitutionsmatrix wird oft dazu verwendet, um einem bestimmten Sequenzalignment eine Score zuzuordnen und damit zu bestimmen, wie gut das Alignment ist. Häufig verwendete Substitutionsmatrizen sind BLOSUM und Point Accepted Mutation Matrix (PAM-Matrix).
Algorithmen wie BLAST oder FASTA verwenden bei der Suche nach ähnlichen Proteinen in einer Datenbank eine Substitutionsmatrix.Inhaltsverzeichnis
Typen von Substitutionsmatrizen
Es gibt verschiedene Arten von Substitutionsmatrizen:
- Einheitsmatrix
- Basierend auf dem genetischen Code
- Basierend auf den chemischen Eigenschaften der Aminosäuren
- Basierend auf empirischen Daten (PAM und BLOSUM, sowie VT, MD BlastP und OPTIMA)
Bei den letzten drei Arten von Matrizen wird berücksichtigt, dass gewisse Mutationen häufiger (wahrscheinlicher) sind als andere. Verbreitet sind aber meist nur Matrizen, die auf empirischen Daten beruhen, wobei die BLOSUM (BLOcks SUbstitution Matrix) und die PAM (Percent accepted Mutations oder Point accepted Mutations)-Matrix am bekanntesten sind.
Einheitsmatrix
Die einfachste Substitutionsmatrix ist die Einheitsmatrix, bei welcher alle nichtidentischen Buchstaben den Wert 0 erhalten und alle identischen Buchstaben den Wert 1. Damit ist die Score dieser Matrix geteilt durch die Länge des Alignments gleich der prozentualen Identität der zwei Sequenzen. Diese Matrix sieht wie folgt aus:e:
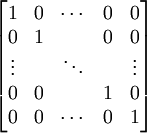
Diese Matrix wäre sehr schlecht, zwei evolutionär weit entfernte Aminosäuresequenzen zu vergleichen. Doch um Nukleidsequenzen (DNA) zu vergleichen, bei der alle Mutationen ähnlich wahrscheinlich sind, wird oft eine solche Matrix verwendet.
Empirische Matrizen
BLOSUM - Matrix
Die BLOSUM Matrizen wurde 1992 von Henikoff und Henikoff berechnet. Es gibt verschiedene Matrizen, die sich nur in den folgenden Zahlen unterscheiden. Die am häufigsten verwendete BLOSUM Matrix ist BLOSUM62. Für die Berechnung der BLOSUM62-Matrix wurden verwandte Proteinsequenzen verglichen, die zu maximal 62% identisch waren. Aus diesem Vergleich geht eine Tabelle hervor, welche die relative Mutationsrate (log odds) darstellt.
PAM - Matrix
Die PAM-Matrix war eine der ersten Aminosäure-Substitutionsmatrizen. Sie wurde in den 1970ern von Margaret Dayhoff entwickelt.
Die Matrix errechnet sich durch die Beobachtung des Unterschieds in nah verwandten Proteinen.
Die PAM1-Matrix gibt an, mit welcher Rate eine Substitution zu erwarten wäre, wenn sich 1% der Aminosäuren verändert hätte, entspricht also einer Ähnlichkeit von 99%. Die höchste Stufe ist PAM250, die einer Sequenzähnlichkeit von ca 20% entspricht, mit höheren Stufen arbeitet man in der Praxis nicht, da man bei einer Wahrscheinlichkeit von unter 20% nicht mehr von Ähnlichkeit sprechen kann.
Die Wahrscheinlichkeiten in einer PAM-Matrix sind der Übersicht halber mit 10000 multipliziert, d.h. in der PAM1 - Matrix unten ist die Wahrscheinlichkeit dafür, dass Glutaminsäure (E) durch Alanin (A) ersetzt wird, gleich 0,0017 oder 0,17%.
Nicht ganz korrekt, aber gut zu merken, ist PAM als Prozentzahl zugelassener Mutationen.
Beispiel einer PAM1 - Matrix
A R N D C Q E G H I L K M F P S T W Y V A 9867 2 9 10 3 8 17 21 2 6 4 2 6 2 22 35 32 0 2 18 R 1 9913 1 0 1 10 0 0 10 3 1 19 4 1 4 6 1 8 0 1 N 4 1 9822 36 0 4 6 6 21 3 1 13 0 1 2 20 9 1 4 1 D 6 0 42 9859 0 6 53 6 4 1 0 3 0 0 1 5 3 0 0 1 C 1 1 0 0 9973 0 0 0 1 1 0 0 0 0 1 5 1 0 3 2 Q 3 9 4 5 0 9876 27 1 23 1 3 6 4 0 6 2 2 0 0 1 E 10 0 7 56 0 35 9865 4 2 3 1 4 1 0 3 4 2 0 1 2 G 21 1 12 11 1 3 7 9935 1 0 1 2 1 1 3 21 3 0 0 5 H 1 8 18 3 1 20 1 0 9912 0 1 1 0 2 3 1 1 1 4 1 I 2 2 3 1 2 1 2 0 0 9872 9 2 12 7 0 1 7 0 1 33 L 3 1 3 0 0 6 1 1 4 22 9947 2 45 13 3 1 3 4 2 15 K 2 37 25 6 0 12 7 2 2 4 1 9926 20 0 3 8 11 0 1 1 M 1 1 0 0 0 2 0 0 0 5 8 4 9874 1 0 1 2 0 0 4 F 1 1 1 0 0 0 0 1 2 8 6 0 4 9946 0 2 1 3 28 0 P 13 5 2 1 1 8 3 2 5 1 2 2 1 1 9926 12 4 0 0 2 S 28 11 34 7 11 4 6 16 2 2 1 7 4 3 17 9840 38 5 2 2 T 22 2 13 4 1 3 2 2 1 11 2 8 6 1 5 32 9871 0 2 9 W 0 2 0 0 0 0 0 0 0 0 0 0 0 1 0 1 0 9976 1 0 Y 1 0 3 0 3 0 1 0 4 1 1 0 0 21 0 1 1 2 9945 1 V 13 2 1 1 3 2 2 3 3 57 11 1 17 1 3 2 10 0 2 9901horizontal: ursprüngliche Aminosäure
vertikal: mutierte AminosäureBeispiel einer PAM250 - Matrix
A R N D C Q E G H I L K M F P S T W Y V A 13 6 9 9 5 8 9 12 6 8 6 7 7 4 11 11 11 2 4 9 R 3 17 4 3 2 5 3 2 6 3 2 9 4 1 4 4 3 7 2 2 N 4 4 6 7 2 5 6 4 6 3 2 5 3 2 4 5 4 2 3 3 D 5 4 8 11 1 7 10 5 6 3 2 5 3 1 4 5 5 1 2 3 C 2 1 1 1 52 1 1 2 2 2 1 1 1 1 2 3 2 1 4 2 Q 3 5 5 6 1 10 7 3 7 2 3 5 3 1 4 3 3 1 2 3 E 5 4 7 11 1 9 12 5 6 3 2 5 3 1 4 5 5 1 2 3 G 12 5 10 10 4 7 9 27 5 5 4 6 5 3 8 11 9 2 3 7 H 2 5 5 4 2 7 4 2 15 2 2 3 2 2 3 3 2 2 3 2 I 3 2 2 2 2 2 2 2 2 10 6 2 6 5 2 3 4 1 3 9 L 6 4 4 3 2 6 4 3 5 15 34 4 20 13 5 4 6 6 7 13 K 6 18 10 8 2 10 8 5 8 5 4 24 9 2 6 8 8 4 3 5 M 1 1 1 1 0 1 1 1 1 2 3 2 6 2 1 1 1 1 1 2 F 2 1 2 1 1 1 1 1 3 5 6 1 4 32 1 2 2 4 20 3 P 7 5 5 4 3 5 4 5 5 3 3 4 3 2 20 6 5 1 2 4 S 9 6 8 7 7 6 7 9 6 5 4 7 5 3 9 10 9 4 4 6 T 8 5 6 6 4 5 5 6 4 6 4 6 5 3 6 8 11 2 3 6 W 0 2 0 0 0 0 0 0 1 0 1 0 0 1 0 1 0 55 1 0 Y 1 1 2 1 3 1 1 1 3 2 2 1 2 15 1 2 2 3 31 2 V 7 4 4 4 4 4 4 4 5 4 15 10 4 10 5 5 5 72 4 17horizontal: ursprüngliche Aminosäure
vertikal: mutierte Aminosäure
Wikimedia Foundation.
