- DNS Sequenzierung
-
DNA-Sequenzierung ist die Bestimmung der DNA-Sequenz, d. h. der Nukleotid-Abfolge in einem DNA-Molekül. Die DNA-Sequenzierung hat die biologischen Wissenschaften revolutioniert und die Ära der Genomforschung (Genomik) eingeleitet. Seit 1995 konnte durch DNA-Sequenzierung das Genom von über 330 verschiedenen Organismen analysiert werden (siehe Sequenzierte Organismen).
Inhaltsverzeichnis
Problemstellung
Die DNA-Sequenzierung als Ablesen der Nukleotidfolge der DNA war über Jahrzehnte hinweg bis in die Mitte der siebziger Jahre ein ungelöstes Problem. Heute stehen verschiedene biochemische bzw. biotechnologische Methoden zur Verfügung, weitere Details dazu im Abschnitt Sequenzierungsmethoden. Die Kunst der DNA-Sequenzierung beschränkt sich jedoch nicht auf diese Methoden des direkten Ablesens: In jeder einzelnen Sequenzierreaktion werden auf Grund technischer Beschränkungen nur kurze DNA-Abschnitte (engl. reads) von weniger als 1000 bp (Basenpaare) abgelesen. In einem Sequenzierprojekt müssen folglich längere DNA-Abschnitte zunächst in kleinere Einheiten zerlegt (Shotgun Sequencing), sequenziert und anschließend wieder zu einer vollständigen Gesamtsequenz zusammengefügt werden. Genomprojekte wie das Humangenomprojekt, bei denen mehrere Milliarden Basenpaare sequenziert werden, erfordern deshalb ein organisiertes und koordiniertes Vorgehen sowie den Einsatz von bioinformatischen Methoden. Um aus den rohen Sequenzdaten biologisch relevante Informationen zu gewinnen (beispielsweise Informationen über vorhandene Gene und deren Kontrollelemente), schließt sich an die Sequenzierung die DNA-Sequenzanalyse an. Ohne sie bleibt jede Sequenzinformation ohne wissenschaftlichen Wert.
Sequenzierungsmethoden
Es gibt heute mehrere Verfahren zum Ablesen der Sequenzinformation von einem DNA-Molekül, noch finden aber überwiegend Weiterentwicklungen der Methode nach Frederick Sanger Verwendung. Die Methode von Maxam und Gilbert ist in erster Linie historisch interessant. Die neuere Pyrosequenzierung, die für Spezialanwendungen genutzt wird, bietet Möglichkeiten der beschleunigten Sequenzierung durch hochparallelen Einsatz.
Maxam und Gilbert Methode
Die Methode von Allan Maxam und Walter Gilbert von 1977 beruht auf der basenspezifischen chemischen Spaltung der DNA durch geeignete Reagenzien und anschließender Auftrennung der Fragmente durch Gelelektrophorese.[1] Die DNA wird zunächst am 5' oder 3'-Ende mit radioaktivem Phosphat oder nicht-radioaktiv (Biotin, Fluorescein) markiert. In vier getrennten Ansätzen werden dann jeweils bestimmte Basen vom Zucker-Phosphat-Rückgrat der DNA modifiziert und abgespalten, beispielsweise wird die Base Guanin (G) durch das Reagenz Dimethylsulfat methyliert und durch Alkalibehandlung mit Piperidin entfernt. Danach wird der DNA-Strang an den jetzt basenlosen Stellen komplett gespalten. In jedem Ansatz entstehen Fragmente unterschiedlicher Länge, deren 3'-Ende stets an bestimmten Basen gespalten worden war. Die Gelelektrophorese trennt die Fragmente nach der Länge auf, wobei Längenunterschiede von einer Base aufgelöst werden. Durch Vergleich der vier Ansätze auf dem Gel lässt sich die Sequenz der DNA ablesen. Ihren Erfindern ermöglichte diese Methode die Bestimmung der Operon-Sequenz eines Bakteriengenoms. Die Methode kommt heute kaum noch zum Einsatz, da sie gefährliche Reagenzien benötigt und schwerer automatisierbar ist als die zur gleichen Zeit entwickelte Didesoxymethode nach Sanger.
Didesoxymethode nach Sanger
Die Didesoxymethode nach Sanger wird auch Kettenabbruch-Synthese genannt und stellt eine enzymatische Methode dar. Sie wurde von Sanger und Coulson um 1975 entwickelt und bereits 1977 mit der ersten vollständigen Sequenzierung eines Genoms (Bakteriophage φX174 [2]) vorgestellt.[3] Sanger erhielt für seine Arbeiten zur DNA-Sequenzierung zusammen mit Gilbert 1980 den Nobelpreis für Chemie.[4]
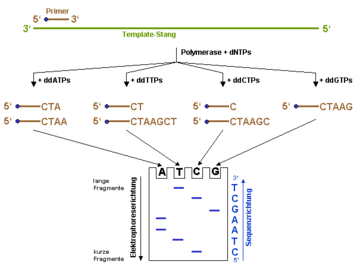 Prinzip der DNA-Sequenzierung nach der Didesoxy-Methode.
Prinzip der DNA-Sequenzierung nach der Didesoxy-Methode.
dNTP ist die allgemeine Abkürzung für ein Nukleosidtriphosphat und kann für dATP, dCTP, dGTP oder dTTP stehen. ddNTPs sind die entsprechenden Didesoxy-Varianten der dNTPs. Der Einbau eines ddNTPs führt zum Abbruch der Polymerisationsreaktion. Die blauen Punkte am 5'-Ende des Primers stellt eine Markierung dar (z.B. eine fluoreszierende Gruppe), mittels der die Syntheseprodukte später im Gel sichtbar gemacht werden können. Alternativ lassen sich auch radioaktiv markierte Nukleosidtriphosphate zur Polymerisationsreaktion einsetzen.Ausgehend von einem kurzen Abschnitt bekannter Sequenz (Primer) wird durch das Enzym DNA-Polymerase einer der beiden komplementären DNA-Stränge verlängert. Zunächst wird die DNA-Doppelhelix durch Erwärmung denaturiert, woraufhin Einzelstränge für das weitere Vorgehen zur Verfügung stehen. In vier sonst gleichen Ansätzen (alle beinhalten die vier Nukleotide) wird je eine der vier Basen zum Teil als Didesoxynukleosidtriphosphat (ddNTP) zugegeben. Diese Kettenabbruch-ddNTPs besitzen keine 3'-Hydroxygruppe: Werden sie in den neusynthetisierten Strang eingebaut, ist eine Verlängerung der DNA durch die DNA-Polymerase nicht mehr möglich, da die OH-Gruppe am 3'-C-Atom für die Verknüpfung mit der Phosphatgruppe des nächsten Nukleotids fehlt. In der Folge entstehen DNA-Fragmente unterschiedlicher Länge, die in jedem Ansatz stets mit dem gleichen ddNTP enden. Entweder der Primer oder die ddNTPs sind radioaktiv markiert. Nach der Sequenzier-Reaktion werden die markierten Abbruchprodukte aus jedem Ansatz mittels Polyacrylamid-Gelelektrophorese der Länge nach aufgetrennt. Durch Vergleich der vier Ansätze kann man die Sequenz nach der Entwicklung des radioaktiven Gels auf einem fotografischen Film ablesen. Die dementsprechend komplementäre Sequenz ist die Sequenz der verwendeten einsträngigen DNA-Matrize. Als Sequenzier-Reaktion kommt heutzutage eine Variation der Polymerase-Kettenreaktion (PCR) zum Einsatz. Anders als bei der PCR wird nur ein Primer eingesetzt, sodass die DNA nur linear amplifiziert wird.
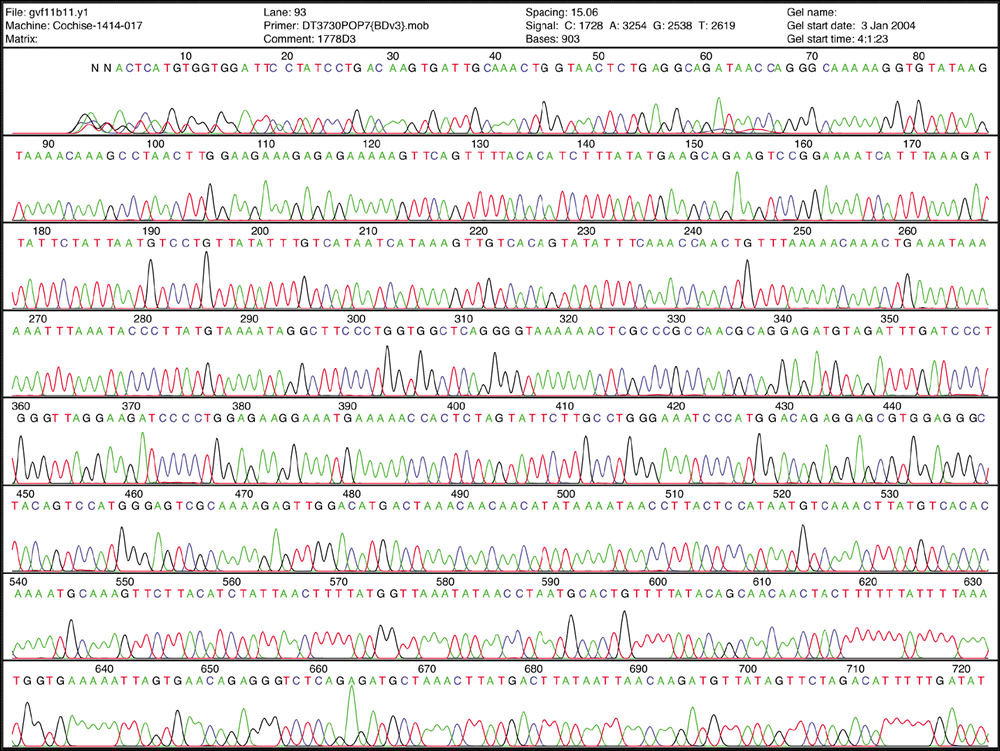
Seit Anfang der neunziger Jahre werden vor allem mit Fluoreszenz-Farbstoffen markierte Didesoxynukleosidtriphosphate eingesetzt. Jedes der vier ddNTPs wird mit einem unterschiedlichen Farbstoff gekoppelt. Diese Modifikation erlaubt es, alle vier ddNTPs in einem Reaktionsgefäß zuzugeben, eine Aufspaltung in getrennte Ansätze und der Umgang mit Radioisotopen entfällt. Die entstehenden Kettenabbruchprodukte werden mittels Kapillarelektrophorese aufgetrennt und mit Hilfe eines Lasers zur Fluoreszenz angeregt. Die ddNTPs am Ende jedes DNA-Fragmentes zeigen dadurch Fluoreszenz unterschiedlicher Farbe und können so von einem Detektor erkannt werden. Das Chromatogramm (die Abfolge der Farbsignale, die am Detektor erscheinen) gibt direkt die Sequenz der Basen des sequenzierten DNA-Stranges wieder.
Pyrosequenzierung
Die Pyrosequenzierung nutzt wie die Sanger-Sequenzierung eine DNA-Polymerase zur Synthese des DNA-Gegenstranges. Allerdings wird die DNA-Polymerase gewissermaßen „in Aktion” beobachtet, wie sie nacheinander einzelne Nukleotide an einen neusynthetisierten DNA-Strang anhängt. Der erfolgreiche Einbau eines Nukleotids wird durch ein ausgeklügeltes Enzymsystem unter Beteiligung einer Luziferase in einen Lichtblitz umgesetzt und von einem Detektor erfasst. Die zu sequenzierende DNA dient als Matrizenstrang und liegt einzelsträngig vor. Ausgehend von einem Primer erfolgt die Strangverlängerung Nukleotid um Nukleotid durch kontrollierte Zugabe von Nukleosidtriphosphaten (NTPs). Bei Zugabe des passenden (komplementären) Nukleotids erhält man ein Signal, bei den unpassenden NTPs bleibt der Lichtblitz aus. Bei Einbau eines komplementären Nukleotids durch die DNA-Polymerase wird Pyrophosphat (PPi) freigesetzt. Das Pyrophosphat wird durch die ATP-Sulfurylase zu Adenosintriphosphat (ATP) umgesetzt. Das ATP treibt die Luziferase-Reaktion an, wodurch Luziferin in Oxyluziferin verwandelt wird. Dies resultiert wiederum in einem detektierbaren Lichtsignal – dessen Stärke proportional zum verbrauchten ATP ist.
Die Pyrosequenzierung wird beispielsweise zur Bestimmung der Häufigkeit von bestimmten Genmutationen (SNPs, engl. Single Nucleotide Polymorphism), z.B. bei der Untersuchung von Erbkrankheiten eingesetzt. Die Pyrosequenzierung ist gut automatisierbar und eignet sich zur hochparallelen Analyse von DNA-Proben.
Sequenzierung durch Hybridisierung
Zu diesem Zweck werden auf einem Glasträger (DNA-Chip oder Microarray) kurze DNA-Abschnitte (Oligonukleotide) in Matrix-Anordnung fixiert. Die Fragmente der zu sequenzierenden DNA werden mit Farbstoffen markiert und das Fragmentgemisch wird auf der Oligonukleotidmatrix ausgebracht, so dass komplementäre fixierte und freie DNA-Abschnitte miteinander hybridisieren können. Nach dem Auswaschen ungebundener Fragmente lässt sich das Hybridisierungsmuster anhand der Farbmarkierungen und deren Stärke ablesen. Da die Sequenzen der fixierten Oligonukleotide und deren Überlappungsbereiche bekannt sind, kann man letztlich aus dem Farbmuster auf die zugrundeliegende Gesamtsequenz der unbekannten DNA rückschließen.
Literatur
Quellen
- ↑ Maxam, A. & Gilbert, W. (1977): A new method of sequencing DNA. In: Proceedings of the National Academy of Sciences U.S.A. Bd. 74, S. 560-564. PMID 265521 doi:10.1038/265687a0PDF
- ↑ Sanger F. et al. (1977): Nucleotide sequence of bacteriophage phi X174 DNA. In: Nature. Bd. 265, S. 687-695. PMID 870828
- ↑ Sanger, F. et al. (1977): DNA sequencing with chain-terminating inhibitors. In: Proceedings of the National Academy of Sciences U.S.A. Bd. 74, S. 5463-5467. PMID 271968 PDF
- ↑ Informationen der Nobelstiftung zur Preisverleihung 1980 an Walter Gilbert und Frederick Sanger (englisch)
Übersichtsartikel
- Laura Bonetta (2006): Genome sequencing in the fast lane. In: Nature Methods. Bd. 3, S. 141-147. PDF doi:10.1038/nmeth0206-141
Hilfsmethoden
- Shotgun Sequencing
- Chromosome Walking
- Pyrosequencing
Weblinks
Zu Maxam-Gilbert- und Sanger-Sequenzierung (deutsch):
Zu Maxam-Gilbert- und Sanger-Sequenzierung (englisch):
Zur Pyrosequenzierung:
- Anschauliche Präsentation des Prinzips (englisch):
- Zum hochparallelen Einsatz (englisch):
Wikimedia Foundation.