- Haem
-
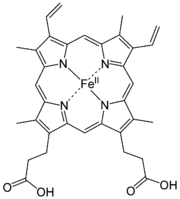
Häme sind Komplexverbindungen mit einem Eisen-Ion als Zentralatom und einem Porphyrin-Molekül als Ligand. Der bekanntester Vertreter ist das Fe-Protoporphyrin IX, auch Häm b genannt, oder einfach Häm. Häme finden sich als prosthetische Gruppe in Proteinen, die als Cytochrome bezeichnet werden.
Inhaltsverzeichnis
Porphyrinstruktur
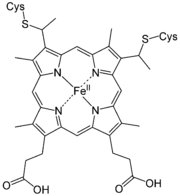
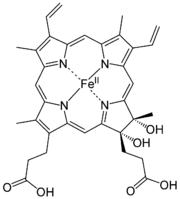
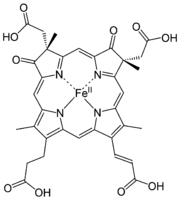
Häme sind eisenhaltige Porphyrinkomplexe. Die Häme unterscheiden sich in der Porphyrinstruktur.
Name Grundstruktur R3 R8 R18 Häm a 
Hydroxyfarnesyl −CH=CH2 −CH=O Häm b −CH=CH2 −CH=CH2 −CH3 Häm c −CH(CH3)SH −CH(CH3)SH −CH3 Häm o Hydroxyfarnesyl −CH=CH2 −CH3 Derivate des Häms
Derivate des Häms (genauer Häm b) unterscheidet man nach der Art der beiden axialen Liganden und der Oxidationsstufe des Eisen-Ions in der jeweiligen Komplex-Verbindung:
Häme und ihre axialen Liganden Häm-Derivat Zentral-Ion axialer Ligand
R1axialer Ligand
R2Häm (Ferrohäm) Fe2+ H2O H2O im Hämoglobin, Myoglobin O2 His (Globin) Hämochrom N-Verbindungen N-Verbindungen Häm (Ferrihäm) Fe3+ H2O H2O im Methämoglobin O2 His (Globin) Hämatin OH− H2O Hämin Cl− H2O Hämichrom N-Verbindungen N-Verbindungen Vorkommen
Häm b kommt z. B. als eisenhaltiger Farbstoff in den roten Blutkörperchen vor. Zusammen mit dem in den Erythrozyten vorkommenden Eiweiß, dem Globin, bildet es Hämoglobin, das für die Sauerstoffaufnahme des Körpers eine zentrale Rolle spielt.
Häme kommen als prosthetische Gruppe in folgenden Proteinen (Cytochromen) vor:
Cytochrome und ihre Häme Cytochrom (Holoprotein) prosthetische Häm-Gruppen
pro Protein (Monomer)Hämoglobin Häm b Myoglobin Häm b Katalase Häm b Succinat Dehydrogenase Häm b Cytochrom c Reduktase 2x Häm b, Häm c Cytochrom c Oxidase 2x Häm a Cytochrom c Peroxidase Häm b Cytochrom P450 Oxidase Häm b Cyclooxygenase Häm b Biosynthese
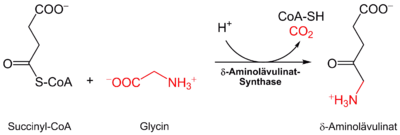
Im menschlichen Körper geht die Biosynthese aller Häme von Succinyl-CoA und der Aminosäure Glycin aus. Die δ-Aminolävulinatsynthase[1] katalysiert die Verbindung von Glycin und Succinyl-CoA zu α-Amino-β-ketoadipat, welches spontan zu δ-Aminolävulinat decarboxyliert:
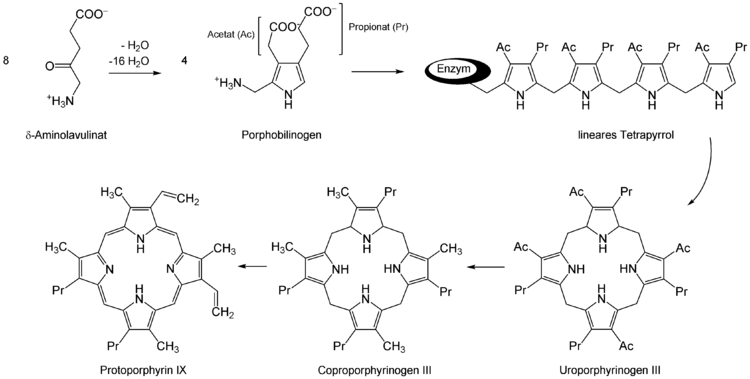
Pflanzen, Algen, Bakterien (mit Ausnahme der Alphaproteobacterien) sowie Archeaebakterien können δ-Aminolävulinat auch ausgehend von Glutaminsäure herstellen, was als C5- bzw. Beale-Weg bezeichnet wird. Dabei wird die an eine tRNA gebundene Aminosäure durch die Glutamyl-tRNA-Reduktase[2] in Glutamat-1-semialdehyd unter NADPH-Verbrauch reduziert. Dieses wird schließlich in δ-Aminolävulinat umgewandelt, was durch die Glutamat-1-semialdehy-aminotransferase katalysiert wird. Es wurde indes noch nicht nachgewiesen, dass Bakterien sowohl den C5-Weg wie auch den oben beschriebenen Syntheseweg über die δ-Aminolävulinatsynthase besitzen [3][4].
Vier dieser Moleküle werden von der PBG-Desaminase unter Abspaltung von vier Ammoniak zu Hydroxymethylbilan umgesetzt. Weitere Umwandlungen erfolgen schrittweise über Uroporphyrinogen III (PBG-Isomerase[5]) und Koproporphyrinogen III (Uroporphyrinogendecarboxylase[6]) zu Protoporphyrinogen IX (Coproporphyrinogen-Oxidase[7]) und Protoporphyrin IX (Protoporphyrinogen-Oxidase[8]). In letzteres baut die Ferrochelatase[9] noch ein zweiwertiges Eisen ein und das Häm (Häm b) ist fertig.
Pathologie: Praktisch jedes der beteiligten Enzyme kann einen Defekt aufweisen, was zu einem typischen Subtyp der Porphyrie führt.Abbau und Ausscheidung
Die Abbauprodukte des Häms heißen Gallenfarbstoffe.
Die Hämooxygenase wandelt das rote Häm noch im Blut unter Sprengung des Porphyrinrings und unter Eisen- und Kohlenmonoxidabspaltung zum grünen Biliverdin um. Die Biliverdinreduktase[10] reduziert es zum orange-roten Bilirubin. Ausgeschieden wird es als Bilirubin mit der Galle oder mit dem Kot.
 Abbau von Häm b (links) zu Bilirubin (rechts).
Abbau von Häm b (links) zu Bilirubin (rechts).Weitere Abbauprodukte des Bilirubins wie das farblose Sterkobilinogen (das durch Darmbakterien zu den braunen Dipyrrolen Mesobilifuchsin und Bilifuchsin umgesetzt wird) und das orange-gelbe Sterkobilin tragen zur normalen Stuhlfarbe bei. Teilweise werden Bilirubinabbauprodukte wieder resorbiert und tragen als Urobilinogen und Urobilin zur gelben Farbe des Urins bei.
Literatur
- P.M. Jordan (1994): Highlights in haem biosynthesis. In: Curr. Opin. Struct. Biol. Bd. 4, S. 902-911. PMID 7712294
- H. Panek, M.R. O'Brian (2002): A whole genome view of prokaryotic haem biosynthesis. In: Microbiology. Bd. 148, S. 2273-2282. PMID 12177321 PDF
- Berg, Tymoczko, Stryer: Biochemie, 5. Auflage, Spektrum Akademischer Verlag GmbH Heidelberg 2003, ISBN 3827413036
Einzelnachweise
- ↑ EC 2.3.1.37 (δ-Aminolävulinatsynthase)
- ↑ EC 1.2.1.70 (Glutamyl-tRNA-Reduktase)
- ↑ H. Panek, M.R. O'Brian (2002): A whole genome view of prokaryotic haem biosynthesis. In: Microbiology. 148, 2273-2282. PMID 12177321 PDF
- ↑ Beale, S.I. (1990): Biosynthesis of the tetrapyrrole pigment precursor, d-aminolevulinic acid, from glutamate. In: Plant Physiology. 93(4), 1273-1279. PMID 16667613 PDF
- ↑ EC 2.5.1.61 (Uroporphyrinogensynthase; PBG-Isomerase)
- ↑ EC 4.1.1.37 (Uroporphyrinogendecarboxylase)
- ↑ EC 1.3.3.3 (Coproporphyrinogen-Oxidase)
- ↑ EC 1.3.3.4 (Protoporphyrinogen-Oxidase)
- ↑ EC 4.99.1.1 (Ferrochelatase)
- ↑ EC 1.3.1.24 (Biliverdinreduktase)
Siehe auch
- Hämine, Hämatine
- Chlorophyll, dort ist das Eisen-Ion des Häms durch ein Magnesium-Ion ersetzt.
- Porphyrien, genetisch bedingte Synthesestörungen der Häm-Gruppe
Weblinks
- PROMISE Datenbank Häm Proteine (engl.)
- Heme and Porphyrin Metabolism (engl.)
- Protopedia: Heme (engl.)
Wikimedia Foundation.