- Regulation der Translation
-
Als Translation wird die Synthese von Proteinen in den Zellen lebender Organismen (siehe auch Proteinbiosynthese) anhand der auf mRNA-Moleküle kopierten genetischen Informationen bezeichnet. Die Translation, als ein wesentlicher Teilprozess der Genexpression, ist der Transkription (komplementäre Kopie der DNA-Informationen auf einzelne mRNA-Stränge) nachgelagert, und erfolgt in lebenden Zellen an besonderen Strukturen, den Ribosomen.
Inhaltsverzeichnis
Allgemeiner Ablauf
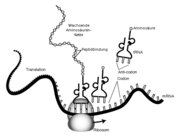 Translation im Cytoplasma; tRNA trägt Aminosäuren diese werden am wachsenden Peptidstrang im Ribosom angehängt.
Translation im Cytoplasma; tRNA trägt Aminosäuren diese werden am wachsenden Peptidstrang im Ribosom angehängt.Die aus der DNA entstandene mRNA enthält die genetische Information für den Aufbau eines Proteins. Diese Information wird nun im Verlauf der Translation genutzt, um das entsprechende Protein zu synthetisieren. Dabei codieren jeweils drei aneinanderfolgende Nukleotide der mRNA (die Codons, oder auch Basentripletts genannt werden) eine bestimmte Aminosäure (siehe: genetischer Code), aus denen das Protein dann sequentiell aufgebaut wird.
Für diesen Vorgang ist als Aminosäuren-„Transporter“ die tRNA notwendig. Diese kann mit ihrem einen Ende (dem sogenannten Anticodon) an je genau einem der Codons auf der mRNA andocken und wurde an ihrem anderen Ende durch die Aminoacyl-tRNA Synthetasen mit der genau zu diesem Codon passenden Aminosäure beladen.
Das Ribosom bringt die mRNA und eine freie tRNA, die eine Aminosäure aufgenommen hat, so zusammen, dass sich an ein bestimmtes Codon auf der mRNA – als passendes Gegenstück – ein komplementäres Anti-Codon der tRNA anlagert. Eine zweite tRNA, die ebenfalls eine Aminosäure trägt, setzt sich neben der ersten tRNA an die mRNA. Die beiden an den tRNAs hängenden Aminosäuren werden mit einer Peptidbindung verknüpft, und die erste tRNA verlässt ohne Aminosäure das Ribosom. Die auf das nächste Codon passende tRNA lagert sich nun an die mRNA an. Ihre Aminosäure wird an die bereits bestehende Aminosäurekette geknüpft und erweitert so diese um ein neues Glied. Dieser Prozess setzt sich fort, so dass sich hinter diesem Punkt eine immer längere werdende Kette aus Aminosäuren bildet. Das Ribosom, das diesen Prozess katalysiert, wandert dabei immer um ein Codon auf der mRNA weiter, und zwar so lange, bis die Information der mRNA vollständig abgearbeitet ist. An dieser Stelle ist ein so genanntes Stopp-Codon (z.B.UGA) in der mRNA eingearbeitet. An dieses kann keine der vorhandenen tRNA-Molekülarten andocken. Hinter der Stelle, an der die Verkettung stattfindet, ist nun eine lange Kette aus Aminosäuren – eine Polypeptidkette – entstanden.
Dieses neu gebildete Eiweiß löst sich nun endgültig vom Ribosom und faltet sich dann meistens so zusammen, dass eine komplexe räumliche Struktur entsteht (Sekundärstruktur und Tertiärstruktur). Eventuell verbindet es sich noch mit anderen Proteinen zu übergeordneten Quartärstrukturen.
Eine mRNA wird in der Regel mehrfach abgelesen, bis sie durch Nucleaseaktivität in ihre Bausteine, Ribonucleotide, zerlegt wird. Bei Eukaryoten ist die Haltbarkeit durch posttranskriptionelle Modifikationen im Kern erhöht.
Siehe auch: Proteomik
Biochemischer Ablauf
Es existieren nur (beim Menschen) 41 verschiedene tRNA-Arten, was nicht äquivalent zu den 64 Triplets ist. Die dritte Position des Codons ist bei vielen Aminosäuren variabel. Hierfür wird die Bezeichnung Wobble-Hypothese verwendet. Die tRNA hat in der Sekundärstruktur die bekannte Kleeblattstruktur, bedingt durch die intramolekulare Basenpaarung komplementärer Nucleotide. Dies entspricht aber nicht der dreidimensionalen Tertiärstruktur, welche eher dem Buchstaben L ähnelt.
Am Akzeptorarm sind die 5'- und 3'-Enden verknüpft. Am 3'-Ende, dem posttransskriptionalem CCA, bindet die entsprechende Aminosäure.
Die Anticodonschleife liegt in der Sekundärstruktur gegenüber, in der Tertiärstruktur hat diese den größtmöglichen Abstand zum Akzeptorarm. Drei zentrale Basen, in der Regel Nummer 34, 35 und 36 bilden das Anticodon. Wobei Position 34 die 1. Base des Anticodons ausmacht, welche mit der 3. Base des Codons paart.
Der D-Arm enthält das ungewöhnliche Dihydrouridin.
Der T-Arm enthält typischerweise Pseudouridin und Cytosin.
Die V-Schleife ist variabel, also bei den tRNAs unterschiedlich zusammengesetzt.
Die Beladung der tRNA mit einer Aminosäure erfolgt durch eine Aminoacyl-tRNA-Synthetase. Für jede Aminosäure gibt es eine spezifische Synthetase. Die genannten Basen gelten im Normalfall, sind aber auch variabel.
Ribosomen und Protein-Synthese
An den Ribosomen erfolgt die Bindung der Aminoacyl-tRNA an die Codons der mRNA und die Synthese der Proteine.
Bei den Ribosomen lassen sich zwei Untereinheiten (jeweils aus RNA und Polypeptiden bestehend) unterscheiden, die zunächst separat existieren. Bei der Translation vereinigen sie sich und bilden zwei funktionell bedeutsame Regionen, an denen sich die tRNAs binden können: an der Peptidyl-Stelle (P-Stelle) sitzt die tRNA mit der wachsenden Proteinkette, an der Aminoacyl-Stelle (A-Stelle) bindet die tRNA mit der nächsten anzufügenden Aminosäure. Eine weitere Region wird als Exit-Stelle (E-Stelle) bezeichnet, an der die entladenen tRNAs das Ribosom verlassen.
Initiation der Translation
Initiationsschritte der TranslationBei der Initiation der Translation (bei Prokaryoten) spielen drei Initiationsfaktoren (IF 1, IF 2, IF 3) eine Rolle.
Die kleine Untereinheit (30S) bildet zu Beginn einen Komplex mit den Initiationsfaktoren 1 und 3[1]. Die Aufgabe des IF3 ist es, eine frühzeitige Bindung der beiden ribosomalen Untereinheiten zu verhindern während IF1 die Dissoziation der (in einem dynamischen Gleichgewicht liegenden) Nichtinitiator-tRNA bezweckt. Der IF2, ein G-Protein, bindet GTP, durchläuft eine Konformationsänderung und erhält dadurch die Möglichkeit die Initiator-fmet-tRNA zu binden. Dieser Komplex hat die Möglichkeit die mRNA und die kleine Untereinheit zu binden. Die Bindung erfolgt durch eine Interaktion der anti-Shine-Dalgarno-Sequenz der 16S-rRNA (ribosomale RNA und Teil der 30S Einheit) mit der Shine-Dalgarno-Sequenz auf der mRNA. Bei dieser handelt es sich um eine upstream (5' vom AUG-Codon gelegen), gelegene, nicht-codierende Sequenz. Dadurch erfolgt auch die Erkennung des Startcodons, durch die Initiator tRNA. Der Abschluss der Initiation wird durch GTP-Hydrolyse am IF2 eingeleitet. Es kommt zum Entlassen der Initiationsfaktoren und zur Bindung der 50S Untereinheit, wodurch der 70S-Initiator-Komplex entsteht. Die fmet-tRNA befindet sich zu Beginn der Translation bereits in der P-Stelle der 50S Untereinheit. Die beiden anderen Stellen, A und E, sind leer.
Elongation der Polypeptidkette
Elongationsschritte der TranslationDie Elongation spielt sich an den A- und P-Orten des Ribosoms ab. (A=Aminoacyl- bzw. Erkennungsort, P=Peptidyl- bzw. Bindungsort). Die E-Stelle (E=Exit, Ausgang) des Ribosoms, dient der Positionierung entladener tRNA.
Die Elongation erfolgt in drei Schritten:
Bindung der Aminoacyl-tRNA in der A-Position
Die Elongation beginnt mit der Bindung einer beladenen tRNA in der A-Stelle. Dieser Vorgang beruht auf dem Transport durch den Elongationsfaktor Tu ([EF-Tu]). Bei diesem handelt es sich wieder um ein G-Protein. EF-Tu hat zwei wichtige Aufgaben: Schutz der Esterbindung zwischen 3-OH der tRNA und der transportierten Aminosäure und Entlassen der tRNA nach Bindung an das Ribosom. Die Entlassung beruht auf der Hydrolyse des gebundenen GTP. Diese kann nur stattfinden bei einer korrekten Paarung zwischen Ribosom und tRNA, also wenn sich die richtige tRNA im Ribosom befindet. Eine falsche tRNA kann nicht vom EF-Tu dissoziieren, wodurch der Einbau der richtigen Aminosäure garantiert wird.
Interessant ist weiterhin zu bemerken, dass EF-Tu jede tRNA erkennen kann, außer der Initiator-tRNA, fmet-tRNA. Somit kann gewährleistet werden, dass bei internen AUG-Codons, nicht fMet, sondern Methionin eingebaut wird.
Die Ausbildung der Peptidbindung
Dies ist ein spontaner Schritt, aufgrund der räumlichen Gegebenheiten der Aminosäuren zueinander und der größeren Bindungsstärke der Peptidbindung gegenüber der Esterbindung (zwischen tRNA und AS). Die katalytische Aktivität hat bei dieser Reaktion die 23S rRNA (!). Das Ribosom ist also ein Ribozym. Durch die Reaktion wird die Bindung des Formyl-Methionins an die Initiator-tRNA gebrochen. Die Initiator-tRNA ist also entladen und es entsteht ein Dipeptid an der zweiten tRNA. Das Peptid bleibt in der P-Stelle der 50S UE (Untereinheit) des Ribosoms, die 30S UE mit mRNA und die beiden tRNAs befinden sich aber noch in ihrer Ausgangsposition.
Der Grund für die Formylierung des Methionins an der Aminogruppe wird nun ersichtlich. Das Dipeptid, welches nach der ersten Elongationsrunde gebildet wird, besitzt die Möglichkeit zum Ringschluss. Der Stickstoff des Methionins hätte die Möglichkeit eines nukleophilen Angriffs auf das Carboxyl-Kohlenstoffatom der zweiten Aminosäure. Es käme zum Ablösen des Dipeptids von der tRNA und somit zur Termination der Elongation.
Wiederherstellung der Ausgangssituation
Die Translokation, Bewegung, der entladenen Initiator-, sowie der zweiten tRNA erfolgt durch den Elongationsfaktor-G. Dieser besitzt eine molekulare Ähnlichkeit zu dem EF-Tu-tRNA-Komplex. Es wird vermutet, dass der EF-G, ähnlich dem EF-Tu an die 50S UE bindet. Der untere Part des EF-G ähnelt einer tRNA, eine Wechselwirkung mit der 30S UE liegt also nahe. Da es sich bei EF-G wiederum um ein G-Protein handelt, unterliegt dieser bei GTP-Hydrolyse einer Konformationsänderung, es verändert also seine Struktur. Dadurch wandert der tRNA-ähnliche Teil Richtung Ribosomeninnenraum und "stößt" die beiden tRNAs eine Position weiter. Die Initiator-tRNA befindet sich nun an der E-Stelle, die zweite tRNA in der P-Stelle. Mit der Dissoziation von EF-G ist der Ausgangszustand wieder erreicht. Eine neue tRNA kann die A-Stelle binden und das Wachstum der Peptidkette vorantreiben.
Termination
Terminationsschritte der TranslationDas Ende der Translation ist erreicht, wenn auf der mRNA eines der Stopp-Tripletts UAG, UAA oder UGA auftaucht. Da es in der Zelle keine passende tRNA für diese Codons gibt, hält die Translation an.
Terminationsfaktoren (release factors) binden an das Stopp-Codon: RF1 an UAG und UAA oder RF2 an UAA und UGA. Ein weiterer RF 3-Faktor vermittelt die Interaktion zwischen RF1/ RF2 und dem Ribosom. Die Spaltung der Bindung zwischen der letzten Aminosäure und der letzten tRNA ist die interessanteste Funktion des Ribosoms. Der Ester kann nicht durch Hydrolyse aufgebrochen werden, da der Bereich der Peptidyl-Transferase vollkommen wasserfrei sein muss, um eine spontane Hydrolyse während der Elongation zu verhindern. Der Mechanismus dieser Reaktion lässt sich durch die Struktur des eukaryotischen Terminationsfaktor erklären. Die Struktur des prokaryotischen RFs ist noch nicht aufgeklärt. Man geht deshalb von einer hypothetischen Homologie aus. Der eukaryotische RF transportiert über eine bestimmte AS-Sequenz genau ein Molekül Wasser in das Peptidyl-Transferase-Zentrum. Dieses kann dann, mit Hilfe katalytischer Aktivität des Ribosoms die Esterbindung spalten. Diese Sequenz befindet sich auch im prokaryotischen RF.
Nun fällt das Protein und die mRNA vom Ribosom ab, das wieder in seine beiden Untereinheiten zerfällt. Der Initiationsfaktor IF 3 erhält den dissoziierten Zustand aufrecht. Somit kann der Kreislauf von Neuem beginnen.
Translation in Eukaryoten
Die Translation in Eukaryoten unterscheidet sich besonders in der Initiation von der prokaryotischen Translation. Es gibt keine fMet-tRNA, sondern eine Initiator-Met-tRNA, welche in vivo nicht formyliert ist. Außerdem gibt es keine Shine-Dalgarno-Sequenz auf der mRNA. Es wird gewöhnlich das erste AUG (5'-seitigste) der mRNA als Startcodon gewählt. Die Bindung der 40S Untereinheit erfolgt an der 5'-Cap-Struktur der mRNA. Daraufhin läuft das Ribosom in 3'-Richtung auf der mRNA entlang und sucht ein AUG. Den Erfolg "der Suche meldet" ein erfolgreiches Paaren der Initiator-Met-tRNA mit der mRNA. Des Weiteren hat die eukaryotische mRNA aufgrund der Prozessierung und des Transports aus dem Kern, genug Zeit komplexe Sekundärstrukturen zu bilden. Diese müssen durch Helicasen wieder aufgebrochen werden.
Translation durch Membranen
Da die meisten Bakterien beziehungsweise Prokaryoten von mehreren stabilen Membranen umgeben sind, haben sich hier einige besondere Mechanismen entwickelt, um Proteine durch Membranen nach außen zu synthetisieren. Auch bei Eukaryoten kommen solche Mechanismen vor, da auch die Organellen von einer (Doppel)-Membran umgeben sind. Es gibt zwei Vorgehensweisen:
Posttranslationaler Proteintransport
Siehe Hauptartikel: Posttranslationaler Proteintransport
Bei diesem Verfahren wird das Protein in der Zelle vollständig zusammengebaut und durch Chaperone wird eine vorzeitige Auffaltung verhindert, wobei bei Bakterien durch einen eingebauten „Knick“ im Protein das Durchfädeln durch die Zellmembran erleichtert wird. Bei Eukaryoten konnte der posttranslationale Transport durch die ER-Membran bisher nur bei Hefen gezeigt werden.
Cotranslationaler Proteintransport
Siehe Hauptartikel: Cotranslationaler Proteintransport
Bei diesem Weg wird das Ribosom durch Signalproteine an die Zellmembran (vor allem des Endoplasmatischen Reticulums) gebracht. Das entstehende Protein wird dann durch einen speziellen Tunnel in den dahinterliegenden Bereich geschoben.
Regulation
Unter Regulation der Translation versteht man verschiedene biochemische Mechanismen, die die Translation steuern. Ein wichtiger Signalweg, der durch Regulation der Translation Zellwachstum und Zellzyklus steuert, ist der mTOR-Signalweg.
Jedes von der Zelle zum Überleben benötigte Protein ist in den Genen kodiert. Die benötigte Menge ist jedoch nicht direkt im Gen enthalten und auch abhängig von Umgebungsbedingungen, Alter und Zellzyklus. Neben der Regulation der Translation hat die Zelle noch einige weitere Möglichkeiten die exprimierte Proteinmenge zu regulieren:
- Regulation der Transkription
Bei eukaryotischen Zellen noch zusätzlich:
- Regulation des Transportes der mRNA aus dem Zellkern
- alternatives Splicing
Die Translation wird ähnlich wie die Transkription reguliert: Ein Repressor bindet an den Translationsstartpunkt und blockiert so die Initiation dieses Prozesses. In einigen Fällen bezieht dies die Erkennung spezieller Strukturen an der mRNA ein.
Beispiel einer Regulation der Translation ribosomaler Proteine
Die korrekte Expression ribosomaler Proteine stellt ein interessantes regulatorisches Problem für die Zelle dar. Jedes Ribosom enthält circa 50 spezielle Proteine, die alle mit derselben Rate synthetisiert werden müssen. Des Weiteren sind die Syntheserate von Proteinen der Zelle und der Bedarf an Ribosomen eng mit dem Zellwachstum verbunden. Eine Veränderung der Wachstumsbedingungen führt schnell zu einem Anstieg oder Absinken der Syntheserate dieser ribosomalen Komponente. Dafür wird eine Regulation benötigt.
Die Kontrolle der ribosomal-Proteingene ist vereinfacht durch die Organisation in verschiedene Operons, die jeweils Gene für bis zu 11 ribosomale Proteine enthält.
Die primäre Kontrolle geschieht auf Ebene der Translation. Dies kann etwa durch das folgende Experiment nachgewiesen werden:
Durch Einbringen zusätzlicher Kopien eines solchen Operons in das Erbgut einer Zelle steigert sich dementsprechend die Menge der durch Transkription erzeugten mRNA. Trotzdem bleibt die Syntheserate des Proteins nahezu unverändert. Die Zelle kompensiert also die erhöhte mRNA Menge. Dabei wirken ribosomale Proteine als Repressoren ihrer eigenen Translation.
Bei jedem Operon kann dabei ein schon synthetisiertes ribosomales Protein an die mRNA des Operons binden. Diese Bindungsstelle liegt in der Nähe eines der ersten Gene des Operons. Dadurch werden Ribosome daran gehindert an die mRNA zu binden und mit der Translation zu beginnen. Die Repression der Translation der ersten Gene verhindert also die Expression von einem Teil oder dem gesamten Rest der nachfolgenden Gene.
Dieser Mechanismus ist sehr empfindlich. Schon wenige nicht zur Bildung von Ribosomen verbrauchte Moleküle des Proteins L4 zum Beispiel verhindern bereits die Synthese dieses Proteins so wie die Synthese der anderen 10 ribosomalen Proteine im gleichen Operon. Dadurch wird also sichergestellt, dass die Proteine nicht in zu großen Mengen erzeugt werden, dass nicht alles zur Bildung von Ribosomen verbraucht werden kann.
Wie ein Protein sowohl als ribosomale Komponente als auch als Regulator seiner eigenen Translation dienen kann, konnte durch Vergleich der Bindungsstellen des Proteins an der rRNA mit den Bindungsstellen mit seiner eigenen mRNA erforscht werden. Diese Bindungsstellen ähneln sich in ihrer Sequenz und ihrer Sekundärstruktur. Da die Bindung der ribosomalen Proteine an die rRNA stärker ist als die an die mRNA, wird die Translation nur unterdrückt, wenn der Bedarf an Proteinen für die Produktion von Ribosomen gedeckt ist.
Literatur
- Rolf Knippers: Molekulare Genetik. 9. Auflage. Thieme, Stuttgart/New York 2006, ISBN 978-3-13-477009-4.
Weblinks
- Lukas Hensel – Animation zur Translation
- Universität Bern – Translation bei Prokaryonten und Eukaryonten
- Zentrale für Unterrichtsmedien im Internet – Translation
- Virtual Cell – Animation einer Translation (englisch)
- IUBMB: Prokaryotic and eukaryotic translation factors
Einzelnachweise
- ↑ James D. Watson; Benjamin Cummings (Hrsg.): Molecular Biology of the Gene. 5 Auflage. ISBN 978-0805346350, S. 435.
Wikimedia Foundation.

