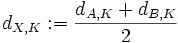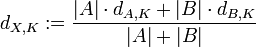Unweighted Pair Group Method
- Unweighted Pair Group Method
-
Unweighted Pair Group Method with Arithmetic mean (kurz UPGMA) bezeichnet eine Bottom-Up Clustering Methode. Sie wird oft in der Bioinformatik zur Rekonstruktion phylogenetischer Bäume angewendet. Im Gegensatz zu anderen Verfahren wie Neighbour Joining basiert UPGMA auf der Annahme der Molekularen Uhr, d.h. dass alle Taxa mit derselben konstanten Rate evolvieren.
Gegeben ist eine Menge von Objekten und eine Distanzmatrix welche die paarweisen Distanzen der Objekte enthält, wobei das Distanzmaß dX,Y die Eigenschaften einer Metrik aufweisen muss. Gesucht ist ein binärer Baum, dessen Blätter die Objekte darstellen und dessen Kanten möglichst gut die Distanzen in der Distanzmatrix reflektieren.
Zu Beginn ist jedes Objekt in einem eigenen Cluster. In jedem Schritt werden die beiden Cluster mit der geringsten Distanz zusammengefasst und die Distanzmatrix neu berechnet. Die Distanz zwischen zwei Clustern ist der Mittelwert der paarweisen Distanzen aller Objekte in beiden Clustern. Sei X der neue Cluster der aus den beiden Clustern A und B gebildet wurde: 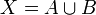 .
.
Die Distanz zu einem Cluster K berechnet sich dann bei WPGMA wie folgt:
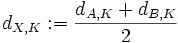
Sind unterschiedlich viele Objekte in einem Cluster, so tragen diese bei WPGMA nicht gleichberechtig zur Abstandsberechnung des neuen Clusters bei. Die Distanzen werden also in der Berechnung unterschiedlich gewichtet (daher: weighted PGMA).
Verwendet man das verbesserte UPGMA, so berechnen sich die neuen Distanzen mit:
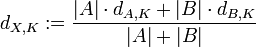
Dies bewirkt, dass alle Abstände gleichberechtigt, also ungewichtet (unweighted), in die Abstandsberechnung einbezogen werden.
Häufig wird WPGMA und UPGMA verwechselt, da die ungewichtete Berechnung, UPGMA, die mathematisch kompliziertere Formel hat, während die Formel gewichtete Berechnung, WPGMA, einfacher aussieht. Bei der Bezeichnung wurde jedoch von dem Ergebnis ausgegangen: Der einfache Mittelwert des WPGMA ergibt ein gewichtetes Ergebnis, während der proportionale Mittelwert des UPGMA ein ungewichtetes Ergebnis liefert.[1]
Literatur
- R.R. Sokal and C.D. Michener.: A statistical method for evaluating systematic relationships. In: University of Kansas Science Bulletin, 38:1409–1438, 1958.
Einzelnachweise
- ↑ http://www.mun.ca/biology/scarr/UPGMA_vs_WPGMA.htm
Wikimedia Foundation.
Schlagen Sie auch in anderen Wörterbüchern nach:
Unweighted Pair Group Method with Arithmetic mean — (kurz UPGMA) bezeichnet eine Bottom Up Clustering Methode. Sie wird oft in der Bioinformatik zur Rekonstruktion phylogenetischer Bäume angewendet. Im Gegensatz zu anderen Verfahren wie Neighbour Joining basiert UPGMA auf der Annahme der… … Deutsch Wikipedia
Unweighted Pair Group Method with Arithmetic Mean — UPGMA (Unweighted Pair Group Method with Arithmetic mean) est le nom d un algorithme destiné à la construction d un arbre phylogénétique. Cette méthode permet la transformation d une matrice de distances (entre différents organismes, populations … Wikipédia en Français
UPGMA — Unweighted Pair Group Method with Arithmetic Mean UPGMA (Unweighted Pair Group Method with Arithmetic mean) est le nom d un algorithme destiné à la construction d un arbre phylogénétique. Cette méthode permet la transformation d une matrice de… … Wikipédia en Français
UPGMA — Unweighted Pair Group Method with Arithmetic mean (kurz UPGMA) bezeichnet eine Bottom Up Clustering Methode. Sie wird oft in der Bioinformatik zur Rekonstruktion phylogenetischer Bäume angewendet. Im Gegensatz zu anderen Verfahren wie Neighbour… … Deutsch Wikipedia
WPGMA — Unweighted Pair Group Method with Arithmetic mean (kurz UPGMA) bezeichnet eine Bottom Up Clustering Methode. Sie wird oft in der Bioinformatik zur Rekonstruktion phylogenetischer Bäume angewendet. Im Gegensatz zu anderen Verfahren wie Neighbour… … Deutsch Wikipedia
UPGMA — unweighted pair group method with averages … Medical dictionary
UPGMA — • unweighted pair group method with averages … Dictionary of medical acronyms & abbreviations
Cluster analysis — The result of a cluster analysis shown as the coloring of the squares into three clusters. Cluster analysis or clustering is the task of assigning a set of objects into groups (called clusters) so that the objects in the same cluster are more… … Wikipedia
UPGMA — (Unweighted Pair Group Method with Arithmetic mean) is a simple agglomerative or bottom up data clustering method used in bioinformatics for the creation of phylogenetic trees. UPGMA assumes a constant rate of evolution (molecular clock… … Wikipedia
Computational phylogenetics — is the application of computational algorithms, methods and programs to phylogenetic analyses. The goal is to assemble a phylogenetic tree representing a hypothesis about the evolutionary ancestry of a set of genes, species, or other taxa. For… … Wikipedia
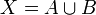 .
.