- High-Throughput Screening
-
High-Throughput-Screening (HTS) ist eine vor allem in der Pharmaforschung angewendete automatisierte Methode, bei der im Hochdurchsatz Zehntausende bis Millionen von biochemischen, genetischen oder pharmakologischen Tests durchgeführt werden. Als Ultra-High-Throughput-Screening (uHTS) werden darüber hinaus Methoden bezeichnet, bei denen mehr als 100.000 Tests pro Tag durchgeführt werden. Das High-Throughput-Screening dient insbesondere der Findung neuer biologisch aktiver Substanzen, die als Leitstruktur für die Entwicklung neuer Arzneistoffe dienen.
Inhaltsverzeichnis
Durchführung
Da beim High-Throughput-Screening große Molekülbibliotheken untersucht werden, werden hohe Anforderungen an die Automatisierung, die Testverfahren und die Auswertung gestellt.
Testverfahren
Beim High-Throughput-Screening zur Entdeckung neuer pharmakologisch aktiver Substanzen finden Target-basierte oder Phänotyp-basierte Testverfahren (Assays) Anwendung.
Target-basiert
Bei Target-basierten Screenings wird die Interaktion der Testsubstanzen mit bestimmten definierten Zielstrukturen (Targets), insbesondere Proteinen, die mit einer Krankheit oder einem physiologischen Prozess in Verbindung gebracht werden, untersucht. Target-basierten Screenings repräsentieren in der pharmazeutischen Industrie die häufigste Form des Screenings von niedermolekularen Substanzen auf eine biologische Aktivität. Sie werden in der Regel in Mikrotiterplatten mit gereinigten oder ungereinigten Proteinen oder indirekt mit Zellen, die das Target-Protein bilden, durchgeführt. Die Interaktion einer Testsubstanz mit dem Target kann direkt in Bindungsassays (in der Regel über die Verdrängung eines markierten Referenzliganden vom Target) oder indirekt über die Beeinflussung der vom Targetprotein aktivierten Signalwege (z. B. Aktivierung von Second Messengern, Protein-Protein-Interaktionen, Protein-Phosphorylierungen und Genaktivierungen) und enzymatischen Reaktionen bestimmt werden. Dazu werden insbesondere biochemische Methoden eingesetzt, bei denen ein Signal als eine Änderungung der Farbintensität, der Fluoreszenz oder der Lumineszenz gemessen wird. Neuere, auf Lumineszenz basierende Verfahren gelten gegenüber photometrischen und fluorimetrischen Methoden als überlegen, da sie weitgehend unempfindlich gegenüber einer Eigenfarbe oder Eigenfluorenzenz von Testsubstanzen sind. Weitere Eigenschaften der Testsubstanzen, wie beispielsweise Löslichkeit und Stabilität spielen eine entscheidende Rolle und können das Testergebnis verfälschen. Auch hochsensitive szintimetrische Methoden, wie beispielsweise Radioligand-Bindungsstudien, werden für das High-Throughput-Screening verwendet.
Phänotyp-basiert
Beim Phänotyp-basierten Screening werden die Effekte von Testsubstanzen auf einen Prozess ohne Definition eines molekularen Targets untersucht. Das Ergebnis des Screenings ist meist von einer Vielzahl an Parametern abhängig. Phänotyp-basierte Screenings werden insbesondere zur Untersuchung von höhermolekularen Verbindungen, wie beispielsweise Proteine, DNA und siRNA, eingesetzt und dienen in der pharmazeutischen Industrie neben der Entdeckung neuer Leitstrukturen meist der Findung eines Targets. Neben Zellen werden hierfür auch Gewebe oder ganze Organismen, wie beispielsweise Fischembryonen, eingesetzt. Der Effekt einer Testsubstanz wird anhand einer phänotypischen Änderung, z. B. der Änderung der Zellform, des Zellwachstums oder der Zellfunktion, beurteilt. Ein Phänotyp-basiertes Screening wird oft mit Hilfe von automatisierten Mikroskopie (High Content Screening) durchgeführt. Limitierend für den Einsatz eines High Content Screenings ist oft der gegenüber Target-basierten Screenings deutlich geringere Durchsatz.
Beurteilung
Da die Testung einer kompletten Molekülbibliothek in einem High-Throughput-Screening oft über mehrere Tage bis mehrere Wochen dauert, ist ein gleichbleibend zuverlässiges Arbeiten des Testverfahrens eine kritische Voraussetzung. Insbesondere bei Verwendung von Zellen muss mit einer Veränderung mit zunehmender Kultivierungszeit gerechnet werden.
Zur Beurteilung der Robustheit der Daten eines High-Throughput-Screenings werden Kontrollsubstanzen untersucht. Im einfachsten Fall umfassen diese einerseits Vehikel oder eine bekanntermaßen inaktive Substanz (Negativkontrolle) und andererseits eine Substanz, die zu einer maximalen Aktivierung oder Inhibition des Test führt (Positivkontrolle). Mit Hilfe des Z'-Faktors
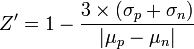 ,
,wobei σp und σn die Standardabweichungen der Positiv- bzw. Negativkontrolle sowie µp und µn die Mittelwerte der Positiv- bzw. Negativkontrolle darstellen, lässt sich das Messfenster des High-Throughput-Screenings beurteilen. Assays mit Z'-Faktoren von mindestens 0,6 gelten als optimal. Abweichend davon werden viele High-Throughput-Screenings auch bei einem Z'-Faktor von 0,3 - 0,6 und seltener auch darunter durchgeführt.
Automatisierung
Der Aufwand eines High-Throughput-Screenings ist nur mit voll- oder zumindest teilautomatisierten Systemen für Liquid-Handling, Datenaufnahme (Reader, Kameras) und gegebenenfalls Zellkultur möglich. Der Einsatz von Mikrotiterplatten mit 384, 1536 oder gar 3456 Wells erlaubt eine Reduzierung des Testvolumens auf wenige Mikroliter und somit eine Kostenersparnis.
Auswertung
Die als Ergebnis des High-Throughput-Screening anfallenden Daten werden statistisch analysiert. Substanzen, die Messwerte jenseits eines bestimmten Schwellenwerts liefern, werden als Treffer („Hits“) eingestuft. Dennoch muss das Auftreten falsch positiver und falsch negativer Ergebnisse berücksichtigt werden. Um die Menge falscher Hits zu reduzieren, wird meist ein zweites, deutlich kleineres Screening durchgeführt, das sich auf die Hits des ersten Screenings beschränkt.
Die aus den Screenings gewonnenen Daten werden auch mit Hilfe von chemoinformatischen Methoden analysiert. Dazu werden die Hits anhand ihrer molekularen Eigenschaften gefiltert. Auf diese Weise können Substanzen, die beispielsweise auf Grund reaktiver Gruppen (z. B. Aldehyde, Michael-Akzeptoren und Nitrogruppen) oder einer Nichterfüllung Lipinskis Rule of Five als ungeeignet für die weitere Entwicklung angesehen werden, von der Leitstrukturkandidatenliste entfernt werden. Schließlich wählt man die vielversprechendsten Hits für die Entwicklung einer Leitstruktur aus.
Beschränkungen
Nur wenige in einem High-Throughput-Screening identifizierten Hits haben die Qualität, als Leitstruktur klassifiziert zu werden. Ein Hit ist somit nicht automatisch eine Leitstruktur und schon gar kein Arzneistoff. Da High-Throughput-Screenings in der Regel bei einer einzigen Testkonzentration mit Hilfe eines einzigen Assays durchgeführt werden, sind quantitative Aussagen über die Wirkstärke (Potenz) und Selektivität der Testsubstanzen nicht möglich. Viele für die Wirksamkeit und therapeutische Sicherheit einer Substanz notwendige pharmakologische Parameter, wie die Zellmembran- und Gewebepermeabilität sowie die Aufnahme, Verteilung, Metabolisierung, Ausscheidung (ADME) und Toxikologie werden in High-Throughput-Assays nicht oder nur unzureichend berücksichtigt. Die Weiterentwicklung von einem Hit bis hin zu einem Arzneistoff und dessen Zulassung dauert in der Regel etwa 10 bis 12 Jahre.
Wikimedia Foundation.

