- Immunopräzipitation
-
Eine Immunpräzipitation (IP, auch Immunopräzipitation genannt) ist eine molekularbiologische Methode, bei der mittels eines Antikörpers ein Antigen aus einer Lösung aufkonzentriert wird. Dieses Antigen kann ein Protein oder ein Peptid sein. In der Regel ist der Antikörper dabei in vitro an ein festes Substrat gekoppelt und bindet über seine Affinität ein spezifisches Antigen in einer Lösung, beispielsweise einem Gewebelysat. Ein bestimmtes Protein wird also aus einem Proteingemisch heraus präzipitiert.Eine Erweiterung dieser Technik ist die sogenannte Ko-Immunpräzipitation, welche zum Nachweis von Protein-Protein-Wechselwirkungen eingesetzt wird. Hier bindet an das Antigen noch zusätzlich dessen Interaktionspartner. So können mit dieser Methode ganze Proteinkomplexe präzipitiert werden.
Ein präzipitiertes Protein und seine Interaktionspartner können im Anschluss mit unterschiedlichen Methoden nachgewiesen werden, beispielsweise mit einem Western Blot oder durch vorherige radioaktive Markierung.
Ablauf
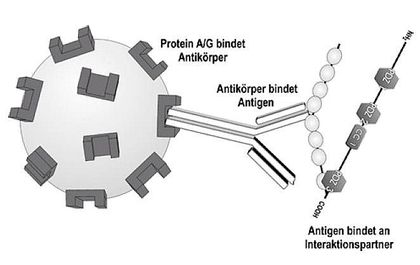 Schema einer Immunpräzipitation. Ein Lysat wird zusammen mit einem spezifischen Antikörper inkubiert. Dieser Antikörper bindet an sein Zielprotein und wird über Protein A/G-Beads präzipitiert. Etwaige Interaktionspartner des Zielproteins, die an dieses gebunden sind, werden mit dem Zielprotein kopräzipitiert. Der Interaktionspartner wird im Western Blot nachgewiesen.
Schema einer Immunpräzipitation. Ein Lysat wird zusammen mit einem spezifischen Antikörper inkubiert. Dieser Antikörper bindet an sein Zielprotein und wird über Protein A/G-Beads präzipitiert. Etwaige Interaktionspartner des Zielproteins, die an dieses gebunden sind, werden mit dem Zielprotein kopräzipitiert. Der Interaktionspartner wird im Western Blot nachgewiesen.Das Proteingemisch kann ein Homogenisat eines Gewebes sein oder aber Zellen aus der Zellkultur. Die Zellkultur ermöglicht es dabei auch, die Partner der vermuteten Interaktion zu überexprimieren, d.h. diese Proteine werden vermehrt gebildet. Der Interaktionspartner sollte in dieser Situation noch an den anderen gebunden sein. Nachdem Zellen oder Gewebe aufgebrochen wurden, gibt man nun Antikörper hinzu, welche an eines der Proteine spezifisch binden. Über diese Antikörper wird dann das gesuchte Protein samt Interaktionspartner herausgezogen. Hierbei bedient man sich in der Regel der spezifischen Eigenschaften von so genanntem Protein A, das aus der Zellwand des Bakteriums Staphylococcus aureus stammt, und/oder Protein G, welches ein Bestandteil der Zellwand von bestimmten Streptokokken-Stämmen ist. Protein A und G binden mit hoher Spezifität an die Fc-Region der meisten Säugetier-Immunglobuline. Mit diesen Proteinen werden nun Kügelchen beschichtet (sogenannte Beads, z.B. aus Sepharose oder magnetischen Mikropartikeln), um in einer solchen Immunpräzipitation die Antikörper-Protein-Komplexe an sich zu binden. Die Isolierung der Komplexe erfolgt nun in der Regel über Zentrifugation sowie mehrere Waschschritte, um unspezifische Proteine zu entfernen. Die Proteine werden von den Beads durch Denaturierung gelöst und der Nachweis erfolgt über einen Western Blot.
Vor- und Nachteile
Eingesetzt wird die IP in der Molekularbiologie als alternativer Nachweis einer Interaktion, z. B. nach einem Hefe-Zwei-Hybrid-Screen. Sie ermöglicht die Untersuchung von Protein-Protein-Interaktionen in zumindest in vivo-ähnlichen Verhältnissen, d. h. im Milieu einer Zelle und mit in Eukaryoten vorkommenden posttranslationale Modifikationen wie Glykosylierung (Anhängen von Zuckerketten), Palmitoylierung (Anhängen von Fettsäuren) oder Faltung durch Chaperone.
Es ist aber möglich, dass sich Proteine durch das Aufbrechen der Zellen verändern oder auch abgebaut werden. Der Erfolg der IP ist auch zu großem Maße von der Bindung des Antikörpers abhängig. Somit können leicht falsch-negative Ergebnisse produziert werden (false negative), die nur durch wiederholte Versuchsserien mit veränderten Bedingungen behoben werden können. Auf der anderen Seite binden manche Proteine auch direkt an die Beads oder an die Oberfläche der Reaktionsgefäße. Diese können eine nicht vorhandene Interaktion vorgaukeln (false positive), welche nur über zusätzliche Kontrollen behoben werden kann.
Des Weiteren ist es möglich, dass zwei Proteine zwar im IP-Versuch interagieren, aber im Zellzyklus, im Zellorganell oder im Zelltyp nicht gleichzeitig auftreten und deshalb nicht tatsächliche Interaktionspartner sein können.
Aus den genannten Gründen muss die Interpretation von IP-Ergebnissen mit Vorsicht erfolgen. Positive Interaktionen müssen immer mit weiteren Techniken aus der Molekularbiologie verifiziert werden, wie beispielsweise Hefe-Zwei-Hybrid-System oder FRET. Das IP-Experimente gibt weiterhin zwar Auskunft über die mögliche Interaktion von zwei Proteinen, jedoch keine Informationen darüber, wie diese Interaktion stattfindet. Dazu sind detailliertere Untersuchungen der Struktur der beteiligten Proteine nötig.
Siehe auch
Wikimedia Foundation.
