- Serin-Protease
-
Serin-endopeptidasen Serine-proteasen (Trypsin, Chymotrypsin und Elastase) Enzymklassifikation EC, Kategorie 3.4.21.- Peptidase Reaktionsart Proteolyse Serinproteinasen sind eine Unterfamilie der Peptidasen (also Enzyme, welche Proteine und Peptide spalten).
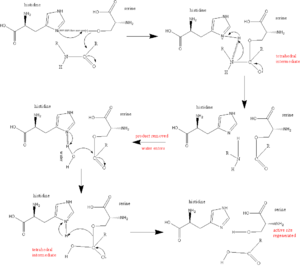
Die Einteilung in diese Gruppe bezieht sich auf das aktive Zentrum der Peptidasen (als aktives Zentrum bezeichnet man den Teil eines Enzyms, der an der Katalyse der chemischen Reaktion beteiligt ist). Wie alle Enzyme haben auch Serinproteasen eine ganz bestimmte Struktur im aktiven Zentrum, hier die katalytische Triade. Bei den Serinproteinasen findet sich dort die Aminosäure Serin.
Zu den Serinproteinasen gehören die Verdauungsenzyme Trypsin, Chymotrypsin, Elastase sowie Plasmin und Thrombin, die beide eine entscheidende Rolle in der Blutgerinnung einnehmen.
Inhaltsverzeichnis
Katalysierte Reaktion
Hemmung
Eine Hemmung von Serinproteinasen findet im Körper beispielsweise durch bestimmte Proteine, die Serpine statt, um die Serinprotease nach getaner Arbeit abzuschalten. Ein Fehlen dieser Inhibitoren führt zu Thrombosen (Beispiel Antithrombin) oder Lungenemphysem (Beispiel Antitrypsin). In solchen Fällen können künstlich hergestellte Hemmstoffe gegeben werden, wie beispielsweise AEBSF und Phenylmethylsulfonylfluorid.
Die Serin-Proteasen unterliegen einer hochdifferenzierten Kontrolle durch ein Netzwerk von miteinander interagierenden Kaskaden von Hemmsubstanzen. Es können zwei Formen unterschieden werden.
Unspezifische Hemmung
Eine Form der Hemmung von Proteasen ist die Umhüllung durch einen Käfig. Das Alpha-2-Makroglobulin umfängt aktivierte Proteasen wie eine Mausefalle. Serin-Proteasen wie Präkallikrein und Thrombin werden so inaktiviert.
Spezifische Hemmung
Ein andere Form der Hemmung von Proteasen ist eine hochspezifische durch Serin-Proteasen-Inhibitoren (Serpine). Viele Enzyme des Gerinnungs-Systems sind solche Serin-Proteasen und Serin-Proteasen-Inhibitoren.
Klassifizierung nach UniProt/MEROPS
Das UniProt-Consortium gibt regelmäßig eine Liste der Peptidasen heraus, die diese Enzyme nach ihrer evolutionären Abstammung kategorisiert. Die Daten der Liste sind, mit hochwertiger Information versehen, in der MEROPS-Datenbank abrufbar. Eng verwandte Moleküle sind dabei in Familien zusammengefasst, deren Bezeichner aus einem Buchstaben ('S' für Serinproteasen) und einer Zahl bestehen. Familien wiederum gehören zu Clans, deren Familien verwandt sind. Clan Bezeichner haben statt Zahlen einen Buchstaben.[1]
Es gibt 45 Serinprotease-Familien in 13 Clans (Stand: 2008), wobei dem Clan PA(S) mit 12 Familien eine herausragende Bedeutung zukommt.[2]
Clan Beschreibung Exemplarisches Enzym UniProt Familien PA(S) Endopeptidasen mit kat. Zentrum His-Asp-Ser Chymotrypsin A (Bos taurus) P00766 S1 S3 S6 S7 S29 S30 S31 S32 S39 S46 S55 S64 PB(S) Autolytische Peptidasen. Nach Autolyse keine Peptidase-Aktivität. Ser am N-Terminus. Penicillin G-Acylase (E. coli) P06875 S45 PC(S) Peptidasen mit kat. Zentrum Ser-His-Glu Dipeptidase E (Salmonella typhimurium) Q1R3S5 S51 SB Subtilase-Familie, Endo- und Exopeptidasen Subtilisin Carlsberg (Bacillus licheniformis) P00780 S8 S53 SC Endo- und Exopeptidasen mit kat. Zentrum Ser-Asp-His Serin-Carboxypeptidase D (Triticum aestivum) P08819 S9 S10 S15 S28 S33 S37 SE Spez. Peptidasen im Metabolismus bakterieller Zellwände D-Ala-D-Ala-Carboxypeptidase B (Streptomyces sp) P15555 S11 S12 S13 SF Endopeptidasen mit kat. Zentrum Ser-His(Lys) UmuD-Protein (E. coli) P0AG11 S24 S26 SH DNA-Virus-Endopeptidasen mit kat. Zentrum His-Ser-His Assemblin (HHV-5) P16753 S21 SJ Endopeptidasen mit kat. Zentrum Ser-Lys Lon-A-Peptidase (E. coli) P0A9M0 S16 S50 S69 SK Endopeptidasen mit kat. Zentrum Ser-His-Asp Peptidase Clp Typ 1 (E. coli) Q1RF98 S14 S41 S49 SP Autopeptidasen mit kat. Zentrum His-X-Ser Nucleoporin 145 (Homo sapiens) P52948 S59 SQ Aminopeptidase DmpA (Ochrobactrum anthropi) S58 SR Endopeptidasen mit kat. Zentrum Lys-Ser Laktoferrin (Homo sapiens) P02788 S60 SS Amidopeptidasen mit kat. Zentrum Ser-Glu-His S66 ST Transmembranpeptidasen mit Transmembrandomänen als Substrat S58 S- ohne Zuordnung S48 S62 S63 S68 S71 S72 Wichtige Serinproteasen
- die Verdauungsenzyme Trypsin, Chymotrypsin, Elastase (S1)
- prostataspezifisches Antigen und Tryptasen (S1)
- die Blutgerinnungsfaktoren Plasmin, Thrombin, Christmas-Faktor, Hageman-Faktor, Faktor VII-aktivierende Protease, Kallikrein (S1) und Stuart-Prower-Faktor (S2)
- MBTPS1 und Proteinase K (S8)
- Dipeptidylpeptidase 4 (S9)
Einzelnachweise
Wikimedia Foundation.