- Snap-Tag
-
Ein Snap-Tag ist ein kommerziell erhältliches System, das es ermöglicht, jedes Protein spezifisch mit einem geeigneten Fluoreszenzfarbstoff zu markieren. Methoden wie diese werden in der Biochemie immer öfter verwendet, um die Funktionsweise von Proteinen und Enzymen in lebenden Zellen zu untersuchen. Gegenüber den aktuellen Standardmethoden, mit denen die Fluoreszenz-Mikroskopie und andere Imaging-Techniken arbeiten, bietet die Verwendung von Snap-Tags deutlich verbesserte Eigenschaften.
Standardmäßig werden Untersuchungen heutzutage mit fluoreszierenden Proteinen, wie z. B. GFP (green fluorescent protein) oder YFP (yellow fluorescent protein) durchgeführt. Die Methoden sind zwar verhältnismäßig leicht durchzuführen, weil die zugrundeliegenden Techniken mittlerweile sehr gut etabliert sind (Bildung von Fusionsproteinen und gezielte Expression in lebenden Zellen) auf der anderen Seite sind die photophysikalischen Eigenschaften die Proteine in der Regel nicht geeignet um damit Einzelmolekülspektroskopie zu betreiben. Sie weisen im Vergleich zu kommerziell erhältlichen Farbstoffe eine sehr viel niedrigere Fluoreszenzquantenausbeute auf und werden durch Anregung mit einem fokussierten Laserstrahl in der Regel schnell zerstört (Photobleaching).
Inhaltsverzeichnis
Funktionsweise
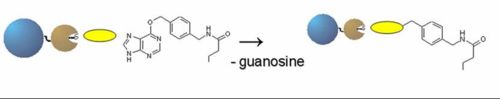
Das sogenannte SNAP-Protein ist ein Derivat eines in Säugetierzellen ubiquitären Enzyms, der O-6-Alkylguanin-alkyltransferase (AGT), die im Organismus die Aufgabe hat, DNA-Defektstellen am Guanosin zu reparieren. Eine ähnliche Funktion hat das SNAP-Protein auch im Verlauf der Markierung des Fusionsproteins. Zur Markierung muss der gewählte Fluoreszenzfarbstoff zunächst an das SNAP-Substrat BG-NH2 gekoppelt werden. Über einen Aminolinker am Substrat wird der NHS-Ester des zu verwendenden Farbstoffs mittels einer SN2-Reaktion kovalent an das Substrat gebunden. Anschließend muss nun das Substrat mit dem Farbstoff durch die Membran hindurch in die Zelle diffundieren. Trifft es dort auf das Snap-Protein, so wird die im Substrat enthaltene Etherfunktion gespalten, das Guanosin wird dadurch freigesetzt und eine direkt kovalente Bindung zwischen dem zu untersuchenden Protein und dem Farbstoff wird ausgebildet.
Reaktionsschema
Quellen und weiterführende Literatur
- Keppler, A. et al. (2004): Labeling of fusion proteins of O6-alkylguanine-DNA alkyltransferase with small molecules in vivo and in vitro. In: Methods. Bd. 32, S. 437-444. PMID 15003606
- Keppler, A. et al. (2004): Labeling of fusion proteins with synthetic fluorophores in live cells. In: Proc. Natl. Acad. Sci. U.S.A. Bd. 101, S. 9955-9959. PMID 15226507
- Juillerat, A. et al. (2005): Engineering substrate specificity of O6-alkylguanine-DNA alkyltransferase for specific protein labeling in living cells. In: Chembiochem. Bd. 6, S. 1263-1269. PMID 15934048
- Brecht, A. & Gibbs, T. (2005): Self Labeling Protein Tags. In: Bioforum. Jg. 2005, Nr. 6, S. 50-51.
Weblinks
Kategorien:- Molekularbiologie
- Zellbiologie
- Biologische Untersuchungsmethode
Wikimedia Foundation.