- Porphobilinogen-Desaminase
-
Porphobilinogen-Desaminase —
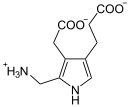
Vorhandene Strukturdaten: 3ecr, 3eq1 Masse/Länge Primärstruktur 361 Aminosäuren Kofaktor Dipyrromethan Isoformen 2 Bezeichner Gen-Name HMBS Externe IDs OMIM: 609806 UniProt: P08397 Enzymklassifikation EC, Kategorie 2.5.1.61 Transferase Substrat 4 Porphobilinogen + H2O Produkte Hydroxymethylbilan + 4 NH3 Vorkommen Homologie-Familie PBG-D Übergeordnetes Taxon Lebewesen Porphobilinogen-Deaminase (PBG-D) (auch: Hydroxymethylbilan-Synthase, HMBS) ist das Enzym, das in allen Lebewesen die Eliminierungsreaktion und anschließende Polymerisation von vier Porphobilinogen-Molekülen zu Hydroxymethylbilan katalysiert. PBG-D ist daher notwendig für die Porphyrin-Biosynthese. Mutationen am HMBS-Gen des Menschen mit folgendem Mangel an PBG-D können zur akuten intermittierenden Porphyrie führen.[1]
Von PBG-D gibt es beim Mensch zwei Isoformen, eine nur in Erythrozyten anzutreffende, die andere wird in den restlichen Gewebetypen exprimiert.
Katalysierte Reaktion
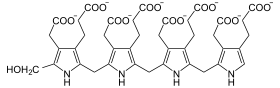
Vier Moleküle Porphobilinogen polymerisieren zu einem Molekül Hydroxymethylbilan, wobei Wasser verbraucht wird und Ammoniak entsteht. Als Kofaktor ist Dipyrromethan notwendig, das gebunden wird und an welches die einzelnen Porphobilinogen-Moleküle zuerst schrittweise addiert werden.[1]
Einzelnachweise
Weblinks
 Wikibooks: Biochemie und Pathobiochemie: Porphyrinbiosynthese – Lern- und Lehrmaterialien
Wikibooks: Biochemie und Pathobiochemie: Porphyrinbiosynthese – Lern- und Lehrmaterialien- Jassal, D'Eustachio / reactome: Four PBGs combine through deamination to form hydroxymethylbilane (HMB)
- OMA: PBG-D Homologe
Wikimedia Foundation.