- Silhouettenkoeffizient
-
Die Silhouette gibt für eine Beobachtung an, wie gut die Zuordnung zu den beiden nächstgelegenen Clustern ist. Der Silhouettenkoeffizient gibt ein von der Cluster-Anzahl unabhängige Maßzahl für die Qualität eines Clusterings an. Der Silhouettenplot visualisiert sowohl alle Silhouetten eines Datensatzes als auch die Silhouettenkoeffizient für die einzelnen Cluster und den Gesamtdatensatz.
Inhaltsverzeichnis
Silhouette
Strukturierung Wertebereich von S(o) stark 
mittel 
schwach 
keine Struktur 
Gehört das Objekt o zum Cluster A so ist die Silhouette von o definiert als:
mit
 die Distanz eines Objekts o zum Cluster A und
die Distanz eines Objekts o zum Cluster A und  die Distanz eines Objekts o zum nächstgelegenen Cluster B. Dabei wird die Differenz des Abstände
die Distanz eines Objekts o zum nächstgelegenen Cluster B. Dabei wird die Differenz des Abstände  gewichtet mit der maximalen Distanz. Damit folgt, dass S(o) für ein Objekt o zwischen −1 und 1 liegt:
gewichtet mit der maximalen Distanz. Damit folgt, dass S(o) für ein Objekt o zwischen −1 und 1 liegt:- Ist die Silhouette S(o) < 0, dann liegen die Objekte des nächstgelegene Clusters B näher an dem Objekt o als die Objekte des Clusters A zu dem das Objekt o gehört. Dies weist darauf hin, dass das Clustering verbessert werden kann.
- Ist die Silhouette
 , dann liegt das Objekt zwischen zwei Clustern und
, dann liegt das Objekt zwischen zwei Clustern und - ist die Silhouette nahe bei Eins, so liegt das Objekt in einem Cluster.
Die Distanz
 wird berechnet als
wird berechnet alsals der Mittelwert der Distanz zwischen allen Objekten im Cluster A und dem Objekt o (nA ist die Anzahl der Objekte im Cluster A). Analog wird die Distanz zum nächstgelegenen Cluster B berechnet als die minimale durchschnittliche Distanz
 .
.
Dabei wird für alle Cluster C, die das Objekt o nicht enthalten, die Distanz
 berechnet. Der nächstgelegene Cluster B ist derjenige, der die kleinste Entfernung
berechnet. Der nächstgelegene Cluster B ist derjenige, der die kleinste Entfernung  aufweist.
aufweist.Silhouettenkoeffizient
Der Silhouettenkoeffizient sC ist definiert als
also als das arithmetische Mittel aller nC Silhouetten des Clusters C definiert. Der Silhouettenkoeffizient kann für jeden Cluster oder für den Gesamtdatensatz berechnet werden.
Beim k-means- oder k-medoid-Algorithmus an kann man mit ihm die Ergebnisse mehrerer Durchläufe des Algorithmus vergleichen um das beste Ergebnis zu behalten. Dies bietet sich insbesondere für die genannten Algorithmen an, da sie randomisiert starten und so unterschiedliche lokale Maxima finden können. Der Einfluss des Parameters k kann so reduziert werden, da der Silhouettenkoeffizient von der Cluster-Anzahl unabhängig ist, und somit Ergebnisse vergleichen kann die mit unterschiedlichen Werten für k erhalten wurden.
Silhouettenplot
Die grafische Darstellung der Silhouetten erfolgt für alle Beobachtungen gemeinsam in einem Silhouettenplot. Für alle Beobachtungen, die zu einem Cluster gehören, wird der Wert der Silhouette als waagerechte (oder senkrechte) Linie dargestellt. Die Beobachtungen in einem Cluster werden dabei nach der Größe der Silhouetten geordnet.
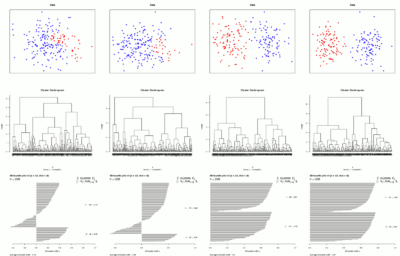
In der rechten Grafiken werden für vier verschiedene Datensätze die Daten, das Dendrogramm für eine hierarchische Clusteranalyse (euklidische Distanz, Single-Linkage) und der Silhouettenplot für die Lösung mit zwei Clustern dargestellt (von oben nach unten). Die Zuordnung der Datenpunkte durch die hierarchische Clusteranalyse in der Zwei-Clusterlösung wird durch die Farben rot (Zuordnung zu Cluster 1) und blau (Zuordnung zu Cluster 2) symbolisiert.
Je besser die beiden Cluster in den Daten getrennt sind (von links nach rechts), desto besser kann die hierarchische Clusteranalyse die Datenpunkte korrekt zuordnen. Auch der Silhouettenplot verändert sich. Während für den linken Datensatz negative Silhouetten vorkommen, finden sich im ganz rechten Datensatz nur positive Silhouetten. Auch die Silhouettenkoeffizienten werden von links nach ganz rechts, sowohl für die einzelnen Cluster als auch für den gesamten Datensatz größer.
Beispiel
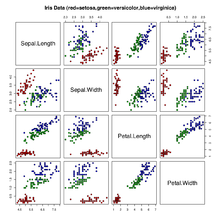
Der Iris flower-Datensatz besteht aus jeweils 50 Beobachtungen dreier Arten von Schwertlilien (Iris Setosa, Iris Virginica und Iris Versicolor), an denen jeweils vier Attribute der Blüten erhoben wurden: Die Länge und die Breite des Sepalum (Kelchblatt) und des Petalum (Kronblatt). Rechts zeigt eine Streudiagramm-Matrix die Daten für die vier Variablen.
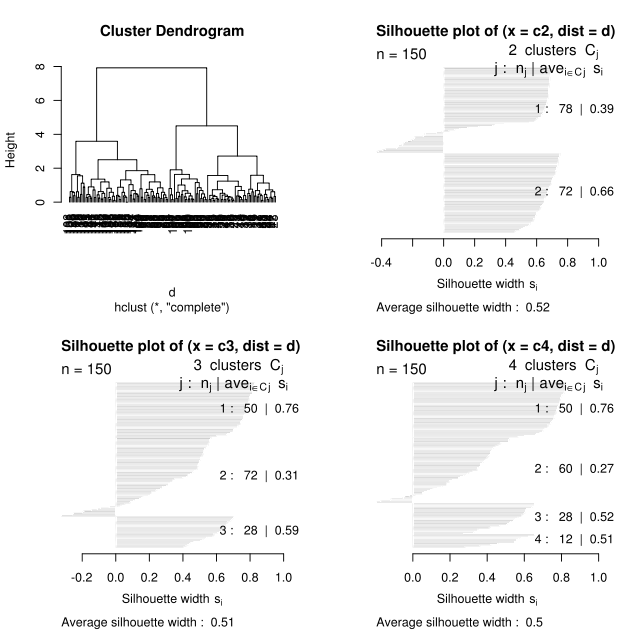
Für die vier Grössen wurde eine hierarchische Clusteranalyse mit der euklidischen Distanz und der Single-Linkage Methode durchgeführt. Oben sind folgenden Grafiken dargestellt:
- Links oben: Ein Dendrogramm der Clusterlösung, hier sieht man das eine zwei oder vier Clusterlösung gut wäre.
- Rechts oben: Grafische Darstellung der Silhouetten der zwei Clusterlösung. Im ersten Cluster sind negative Silhouetten S(o) zu finden, so dass diese Beobachtungen eher falsch zugeordnet sind. Eventuell ist eine Lösung mit mehr Clustern besser geeignet.
- Links unten: Grafische Darstellung der Silhouetten der drei Clusterlösung. Der erste Cluster in wird in zwei Subcluster zerlegt (78 = 50 + 28); zwar sind im ersten Cluster die negativen Silhouetten verschwunden, jedoch haben Beobachtungen im zweiten Cluster nun negative Silhouetten.
- Rechts unten: Grafische Darstellung der Silhouetten der vier Clusterlösung. Der zweite Cluster von der zwei Clusterlösung wird nun in zwei Teilcluste aufgeteilt (72 = 60 + 12), es gibt fast keine negativen Silhouetten mehr.
Es ergeben sich folgende Silhouettenkoeffizienten
Silhouettenkoeffizienten
nC / sCAnzahl Cluster Total 2 150 / 0,52 78 / 0,39 72 / 0,66 3 150 / 0,51 50 / 0,76 28 / 0,59 72 / 0,31 4 150 / 0,50 50 / 0,76 28 / 0,52 60 / 0,27 12 / 0,51 Literatur
- Martin Ester, Jörg Sander: Knowledge Discovery in Databases: Techniken und Anwendungen. Springer, Hamburg/Berlin 2000, ISBN 3-540-67328-8, S. 66. Online: Eingeschränkte Vorschau in der Google Buchsuche
- Peter J. Rousseeuw: Silhouettes: a Graphical Aid to the Interpretation and Validation of Cluster Analysis. In: Computational and Applied Mathematics. 20, 1987, S. 53–65. doi:10.1016/0377-0427(87)90125-7.
Weblinks
- silhouette: Berechnen von Silhouettenkoeffizienten und -plots mit R.
Wikimedia Foundation.