- J-Index
-
Der Balaban-J-Index[1] ist ein topologischer Index, der sich dadurch auszeichnet, auch ähnliche Molekülstrukturen unterscheiden zu können. Hierbei wird von einer geringen Neigung zur Degenerierung gesprochen. Der J-Index wird aus der Distanzmatrix der Molekülstruktur bestimmt, wobei Wasserstoffe nicht berücksichtigt werden.
Inhaltsverzeichnis
Gleichung
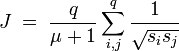
mit
q: Anzahl der Bindungen im Molekül
μ: Anzahl der Ringe im Molekül
- mit
- n: Anzahl der Atome im Molekül
si und sj: Summe der gewichteten Distanzen zwischen dem Atom i bzw. dem Atom j und allen anderen Atomen des Moleküls. Die Distanz ist dabei die Anzahl der Bindungen zwischen den Atomen. In Fällen mehrerer möglicher Wege (durch Ringe) muss der kürzeste Wege gewählt werden. Einzelbindungen gehen mit dem Faktor 1 ein, Doppelbindungen mit dem Faktor 1/2, Dreifachbindungen mit dem Faktor 1/3 und aromatische Bindungen mit dem Faktor 2/3.
Für die Summe
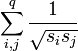 werden nur verbundene (benachbarte) Atome berücksichtigt.
werden nur verbundene (benachbarte) Atome berücksichtigt.Beispielrechnung
Die Distanzmatrix ist eine Tabelle, die die Anzahl der Bindungen zwischen zwei Atomen des Moleküls verzeichnet:
Atom 1 2 3 4 5 6 7 8 Distanzsumme
si1 0 1 2 3 4 5 3 4 22 2 1 0 1 2 3 4 2 3 16 3 2 1 0 1 2 3 1 2 12 4 3 2 1 0 1 2 2 3 14 5 4 3 2 1 0 1 3 4 18 6 5 4 3 2 1 0 4 5 25 7 3 2 1 2 3 4 0 1 16 8 4 3 2 3 4 5 1 0 22 - Anzahl der Bindungen ist q=7
- Anzahl der Atome ist n=8
- Anzahl der Ringe ist μ=0 (=7-8+1)
Die Summe ergibt sich aus
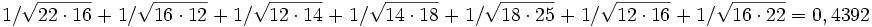
Damit ergibt sich der Balaban-J-Index für Ethylhexan zu
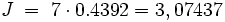 (nicht gerundet)
(nicht gerundet)Bedeutung
Der J-Index zeichnet sich dadurch aus, dass er eine sehr detaillierte Unterscheidung auch ähnlicher Strukturen erlaubt. Durch diese hohe Fähigkeit zur Unterscheidung kann der J-Index erfolgreich bei QSPR-Verfahren (quantitative Eigenschafts-Struktur-Beziehung) (QSPR) verwendet werden.
Literatur
- ↑ Balaban A.T., „Highly Discriminating Distance-based Topological Index“, Chem.Phys.Lett., 89(5), 399-404, 1982
Wikimedia Foundation.

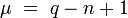
.PNG)