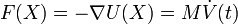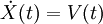- Molekulardynamik
-
Moleküldynamik oder Molekulardynamik (MD) ist eine Art Computersimulation , bei der Atome und Moleküle für einen gewissen Zeitraum unter bekannten Naturgesetzen wechselwirken. Im Allgemeinen bestehen molekulare Systeme aus einer Vielzahl von Partikeln. Es ist daher unmöglich, die Eigenschaften solcher komplexen Systeme auf analytische Weise zu berechnen. MD Simulation benutzt statt dessen einen numerischen Ansatz und ist somit als Schnittstelle zwischen Laborexperimenten und Theorie anzusehen.
Der Begriff Moleküldynamik wird manchmal auch als Synonym für die Discrete element method (DEM) gebraucht, weil die Methoden sehr ähnlich sind. Die Partikel in DEM müssen aber keine chemische Moleküle sein. Im Allgemeinen steht der Begriff MD für die Simulation in verschiedensten Bereichen der Chemie (Anorganische, Organische, Physikalische, Theoretische und Biochemie) sowie angrenzender Gebiete (Materialwissenschaften, Biologie, Pharmazie, Medizin).
Physikalische Prinzipien
Mikrokanonisches Ensemble (NVE)
Das System ist isoliert und tauscht keine Partikel (N), Volumen (V) oder Energie (E) mit der Umgebung aus. Für ein System mit N Partikeln, mit zugehörigen Koordinaten X und Geschwindigkeiten V, kann man folgendes Paar gewöhnlicher Differentialgleichungen aufstellen:
Die Funktion der potenziellen Energie U(X) beschreibt dabei die gegenseitigen Wechselwirkung der Atome und Moleküle. U(X) ist auch als Kraftfeld bekannt. Dieses wird durch zwei Teile definiert: Die mathematische Form (d.h. der funktionale Ansatz für die einzelnen Wechselwirkungsarten, meist der Klassische Mechanik entlehnt) einerseits und die atomspezifischen Parameter andererseits. Letztere erhält man aus spektroskopischen Experimenten und/oder quantenmechanischen Berechnungen (Quantenchemie). Es kann daher für einen Kraftfeldansatz verschiedene Parametersätze geben, z.B. wenn Parameterverfeinerungen durchgeführt wurden. Die Parametrisierung eines Kraftfeldes mit einem weitgespannten Anwendungsbereich stellt damit im Allgemeinen eine große Herausforderung dar.
Bei der Durchführung von MD-Simulationen ist die Wahl des richtigen Kraftfeldes eine wichtige Entscheidung. Generell sind Kraftfelder immer nur für bestimmte Systeme anwendbar (z.B. Proteine oder Silikate), für die sie parametrisiert sind.
Wikimedia Foundation.