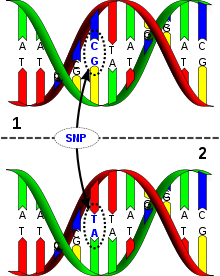
- Single Nucleotide Polymorphism
-
Mit Einzelnukleotid-Polymorphismen (SNP, engl. Single Nucleotide Polymorphism; sprich: Snip) werden Variationen einzelner Basenpaare in einem DNA-Strang bezeichnet. Die Definition, dass SNPs bei mindestens 1 % der jeweiligen Population vorkommen müssen, ist nach der Einführung neuester molekulargenetischer Methoden in der Praxis nicht mehr relevant.
SNPs stellen ca. 90 % aller genetischen Varianten im menschlichen Genom dar; sie treten nicht gleichverteilt auf, sondern nur ungleichmäßig stark an bestimmten Regionen. Zwei Drittel aller SNPs bestehen aus dem Austausch von Cytosin durch Thymin, da Cytosin im Wirbeltier-Genom häufig methyliert wird. Durch spontan auftretende Desaminierung wird aus 5-Methylcytosin Thymin. Je nach Basenaustausch kann die Information des Codon verändert sein. Codiert das Triplett weiterhin für die gleiche Aminosäure spricht man von einem synonymen SNPs. Bei einem nicht-synonymen SNP erfolgte ein Aminosäurewechsel. Liegt ein nicht-synonymer SNP in einer codierenden Region, cSNP (codingSNP) kann eine veränderte Base eine Auswirkung auf die Proteinfunktion haben und physiologische Defekte nach sich ziehen. Eine ebenfalls große Gruppe an SNPs stören zum einen die Genregulation, rSNP (regulatorySNP). Diese liegen häufig in regulatorischen Regionen und beeinflussen die Transkription und somit die Proteinkonzentration. Zum anderen kann die RNA-Prozessierung von srSNP (structural RNASNP) gestört werden. Z.B. kann eine Basenänderung an einer Spleißstelle zu einer mRNA führen, die für ein funktionsloses Protein codiert.
SNPs werden im allgemeinen als "erfolgreiche" Punktmutationen bezeichnet, d. h. als genetische Veränderungen, die sich zu einem gewissen Grad im Genpool einer Population durchgesetzt haben. Einige SNPs korrelieren z. B. mit bestimmten Reaktionen des Organismus bei bestimmten Infektionen oder Kontakt mit speziellen Substanzen.
Ihre wissenschaftliche Bedeutung liegt im häufigen Auftreten und der hohen Variabilität, außerdem sind sie sehr schnell und einfach zu bestimmen. Deswegen werden sie zum Beispiel bei der Suche nach Quantitative Trait Loci, also Chromosomenabschnitten mit Einfluss auf die Ausprägung eines quantitativen Merkmals, zur Identifikation von Individuen und bei Verwandtschaftsdiagnosen, aber auch in der Forschung zur Medikamentenverträglichkeit u. ä. genutzt.
Siehe auch
Weblinks
Wikimedia Foundation.