- Basenpaar
-
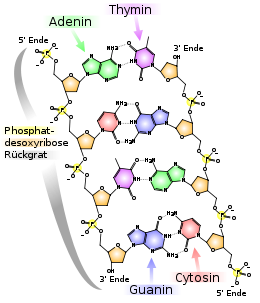
Als Basenpaar bezeichnet man zwei Nukleobasen in der DNA oder RNA, die zueinander komplementär sind und durch Wasserstoffbrückenbindungen zusammengehalten werden. Die Anzahl der Basenpaare eines Gens stellt ein wichtiges Maß der Information dar, die im Gen gespeichert ist. Sie wird in
- bp = Basenpaaren und
- kbp oder kb = Kilo-Basenpaaren (1000 Basenpaaren) gemessen.
Die Längen für größere DNA-Abschnitte werden auch in
- Mbp oder Mb = Megabasenpaaren angegeben.
Ein bp entspricht einer Informationsmenge von 2 Bit, da es vier verschiedene Werte darstellen kann.
Inhaltsverzeichnis
Bedeutung der Basenpaare
Die Basenpaarung spielt eine wesentliche Rolle für die DNA-Replikation, für die Transkription und Translation im Zuge der Proteinbiosynthese sowie für vielfältige Ausgestaltungen der Tertiärstruktur von Nukleinsäuren.
- Replikation: Die neuen Nukleotid-Stränge erhalten durch die Basenpaarung die komplementäre Basensequenz zu den beiden Vorlagensträngen.
- Transkription: Die mRNA, tRNA und rRNA erhält durch die Basenpaarung die zum codogenen Strang der DNA komplementäre Basensequenz, wobei A mit U gepaart wird.
- Die drei Basen des Anticodons der tRNAs paaren sich mit den komplementären Basentriplett der mRNA. Dadurch entsteht die von der DNA codierte Aminosäuresequenz der Eiweiße. Hier treten auch die Wobble-Paarungen bei der Paarung der 3. Base eines Codons der mRNA mit der 1. Base der tRNA auf.
Paarungsregeln
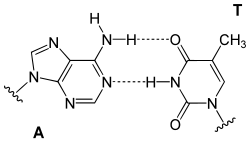
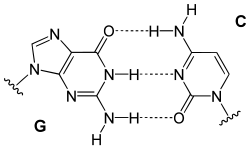
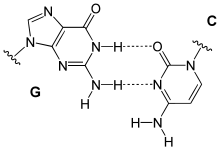
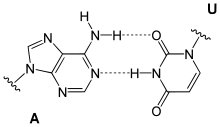
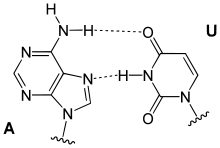
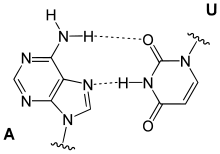
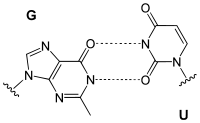
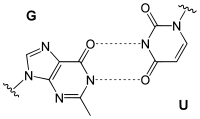
Grundsätzlich bilden immer jeweils genau eine Purin-Base (Guanin oder Adenin) mit einer Pyrimidin-Base (Cytosin oder Thymin bzw. Uracil) ein Basenpaar. Daraus ergeben sich folgende Paarungen:
Watson-Crick-Paarungen
Bereits 1949 stellte der österreichische Biochemiker Erwin Chargaff fest, dass in der DNA die Anzahl der Basen Adenin (A) und Thymin (T) stets im Verhältnis 1 : 1 vorliegt, ebenso beträgt das Verhältnis der Basen Guanin (G) und Cytosin (C) 1 : 1. Dagegen variiert das Mengenverhältnis A : G beziehungsweise C : T stark (Chargaff´sche Regeln).
Daraus schlossen James D. Watson und Francis Harry Compton Crick, dass A-T und G-C jeweils komplementäre Basenpaare bilden.
In der tRNA und rRNA treten ebenfalls Basenpaarungen auf, wenn der Nukleotid-Strang Schleifen bildet und sich dadurch komplementäre Basensequenzen gegenüber stehen. Da in der RNA statt Thymin nur Uracil eingebaut wird, sind die Paarungen A-U und G-C.
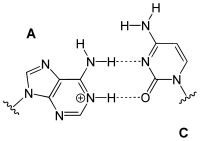
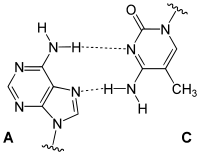
Ungewöhnliche Paarungen
Ungewöhnliche Paarungen treten vor allem in tRNAs und in Tripelhelices auf. Sie folgen zwar dem Watson-Crick-Schema, bilden aber andere Wasserstoffbrückenbindungen aus: Beispiele sind Reverse-Watson-Crick-Paarungen, Hoogsteen-Paarungen (benannt nach Karst Hoogsteen, geboren 1923) und Reverse-Hoogsteen-Paarungen
Wobble-Paarungen
Die Bezeichnung bezieht sich auf die Wobble-Hypothese von Francis Crick (1966). Wobble-Paarungen sind die nicht Watson-Crick-Paarungen G-U oder G-T und A-C:
Siehe auch
Weblinks
- Eine große Datenbank mit zahlreichen Strukturen von Basenpaaren (Abteilung Biologie und Biochemie, University of Houston)
Wikimedia Foundation.