- Dinitrogenase
-
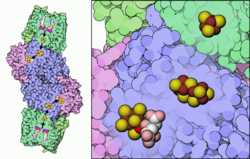
Nitrogenase Links: Kalottenmodell der Nitrogenase von Azotobacter vinelandii (stabilisiert) nach PDB 1N2C. Fremdmoleküle und Elektronenfluss hervorgehoben. Rechts: Detail mit Fe-S- und Fe-S-Mo-Homocitrat-Cluster, das katalytische Zentrum. Bezeichner Gen-Name(n) nif, anf, vnf Enzymklassifikation EC, Kategorie 1.18.6.1 Oxidoreduktase Substrat reduziertes Ferredoxin + H+ + N2 + ATP + H2O Produkte oxidiertes Ferredoxin + H2 + NH3 + ADP + Phosphat Vorkommen Übergeordnetes Taxon Bakterien, Archaeen Nitrogenase ist ein Enzymkomplex, der in der Lage ist, elementaren, molekularen Stickstoff (N2 Dinitrogen) zu reduzieren und damit in eine biologisch verfügbare Form umzuwandeln. Diesen Vorgang bezeichnet man als Stickstofffixierung. Der Enzymkomplex besteht aus zwei Proteinen, der Dinitrogenase und der Dinitrogenase-Reduktase. Nitrogenasen sind bei verschiedenen Bakterien und einigen Archaeen vorhanden. Stickstofffixierende Aktinomyceten sind ebenso bekannt wie Cyanobakterien (zum Beispiel Anabaena) und Proteobakterien (zum Beispiel Azotobacter).
Inhaltsverzeichnis
Struktur und Eigenschaften
Das aktive Zentrum des Enzyms besteht aus einem Eisen-Schwefel-Cluster mit Molybdänatomen. Einige Bakterien sind in der Lage, bei Molybdänmangel alternative Nitrogenasen herzustellen, die statt Molybdän Vanadium oder nur Eisen enthalten.[1][2]
Die Nitrogenase der meisten Bakterien ist extrem sauerstoffempfindlich. Um das Enzym gegen Sauerstoff zu schützen, haben Bakterien verschiedene Anpassungen entwickelt, etwa dicke Schleimkapseln oder besonders dickwandige Zellen. Bakterien, die oxygene Photosynthese betreiben, trennen stickstofffixierende Zellen (Heterocysten) räumlich von Sauerstoff freisetzenden Zellen oder sie assimilieren Stickstoff nur nachts, wenn die Lichtreaktion der Photosynthese ruht. Einige Bakterien können Stickstoff nur in Symbiose fixieren, zum Beispiel Rhizobien (Knöllchenbakterien), die mit Pflanzen (oft Leguminosen) zusammenleben. Da die Pflanzen selbst nicht in der Lage sind, elementaren, molekularen Stickstoff zu fixieren, sind sie auf das Produkt der bakteriellen Nitrogenase mit angewiesen. Das gebildete Ammoniak ist Ausgangsstoff für die Bildung von Glutaminsäure und Glutamin.
Streptomyces thermoautotrophicus, ein thermophiles Bakterium, das aus Kohlenmeilern isoliert wurde, besitzt eine ungewöhnliche, sauerstoffunempfindliche Nitrogenase.
Katalysierte Reaktion
Da N2 ein sehr stabiles und inertes Molekül ist (die Bindungsenergie beträgt 945 kJ/mol), wird eine große Menge an Energie in Form von ATP benötigt, um die Dreifachbindung zwischen den beiden Stickstoff-Atomen zu spalten. Als Produkt dieser Reaktion entsteht Ammoniak.
Die Summengleichung der durch Nitrogenase katalysierten Reaktion lautet:
- N2 + 8 H+ + 8 e- + 16 ATP → 2 NH3 + H2 + 16 ADP + 16 Pi
Die Aktivität der Nitrogenase ist nicht auf Stickstoff beschränkt, das Enzym reduziert auch andere Dreifachbindungen, zum Beispiel die von Ethin oder Cyanid. Unter natürlichen Bedingungen hat diese Eigenschaft vermutlich keine Bedeutung. Die Reduktion von Ethin zu Ethen wird jedoch genutzt, um Nitrogenase experimentell nachzuweisen.
Regulation
Der gesamte Prozess der biologischen Stickstofffixierung ist relativ komplex und erfordert das Zusammenwirken mehrerer Enzyme, von denen die Nitrogenase das wichtigste ist. Die Gene dieser Enzyme unterliegen einer strengen Regulation. Ihre Transkription wird zum Beispiel durch Sauerstoff und Stickstoffverbindungen wie Nitrat und einige Aminosäuren abgeschaltet, was vorteilhaft ist, weil die Nutzung von Stickstoff aus diesen Quellen wesentlich weniger Energie verbraucht.
Siehe auch
Einzelnachweise
Wikimedia Foundation.