- Mehrsubstratreaktion
-
Die Behandlung von Mehrsubstratreaktionen ist ein wichtiges (aber vernachlässigtes) Teilgebiet der Enzymkinetik.
Enzyme arbeiten im Normalfall mit mehreren Substraten, die zu einem oder mehreren Produkten umgesetzt werden. Die Umsetzung nur eines Substrates ist die absolute Ausnahme (Beispiel: Hydrolase- und Isomerasereaktionen). Dennoch bezieht sich die zentrale Gleichung der Enzymkinetik, die Michaelis-Menten Beziehung, – strikt genommen – auf diesen Ausnahmefall.
Als Prototyp einer enzymatischen Umsetzung soll hier die Reaktion der Lactatdehydrogenase (LDH) besprochen werden, das heißt die Umsetzung des Pyruvats mit NADH,H+ zu Lactat und NAD+ (Kopfzeile in Abb. 1), wobei Pyruvat (B) und NADH,H+ (A) die Rolle der beiden (Co-)Substrate und Lactat (Q) bzw. NAD+ (P) die Rolle der beiden Produkte spielen.
Steht man vor der Aufgabe, die enzymkinetischen Parameter (Michaeliskonstanten oder Km-Werte genannt) für beide Substrate zu ermitteln, so ist das klassische Vorgehen das folgende:
- der Km-Wert für Pyruvat ergibt sich, indem die Pyruvatkonzentration in Gegenwart eines großen Überschusses (Sättigungskonzentration) des Zweitsubstrates NADH,H+ variiert wird;
- der Km-Wert für NADH,H+ wird entsprechend bestimmt, indem die NADH,H+-Konzentration in Gegenwart eines sättigenden Überschusses von Pyruvat ermittelt wird.

Abb. 1: Mehrsubstratreaktion am Beispiel der LDH-Reaktion: Variation beider Substrate A: Substratumsatz von Pyruvat durch LDH bei drei nicht-sättigenden Konzentrationen von NADH,H+ (A3>A2>A1). Im Lineweaver-Burk Diagramm entsteht scheinbar das Muster einer nichtkompetitiven Inhibition. B: sekundäre Auftragung, in der die Achsenabschnitte (Rauten)aus Teil A zur Bestimmung des Km-Wertes für das zweite Substrat (NADH,H+) dienen
Eine Alternative ist die Variation beider Substrate nach dem in Abb. 1 gezeigten Schema, das heißt die schrittweise Erhöhung der Pyruvat-Konzentration in Gegenwart einer jeweils anderen, nicht-sättigenden Konzentration von NADH,H+. Ein solches Vorgehen hat den speziellen Vorteil, dass es zusätzlich Informationen über den Reaktionsmechanismus liefert. Damit betreten wir die „hohe Schule der Enzymkinetik“ – gewiss ein Abschnitt der schwierig zu vermitteln ist und auch kaum mehr vermittelt wird. Wenn dieser Versuch hier dennoch unternommen wird, dann um den klassischen Weg aufzuzeigen, auf dem enzymatische Grundfunktionen aufgeklärt werden können und aufgeklärt wurden. Auch heute noch lassen sich derartige Erkenntnisse durch moderne Verfahren (Strukturanalysen) zwar untermauern, aber kaum ersetzen.
Inhaltsverzeichnis
Sequenzielle und Ping-pong Mechanismen
Die Lactatdehydrogenase arbeitet nach einem „sequenziellen“ Mechanismus. Dies bedeutet, dass alle Substrate an das Enzym binden müssen, bevor die Reaktion stattfindet und die Produkte freigesetzt werden. Da hier zwei Substrate (A und B) zu zwei Produkten (P und Q) werden, spricht man (in der Nomenklatur von W. Wallace Cleland) auch von einem „sequenziellen bi-bi“ Mechanismus (Abb. 2A).
Andere Enzyme, wie zum Beispiel die Aminosäure-Transaminasen, bedienen sich hingegen eines „Ping-Pong-bi-bi“ Mechanismus, in dem ebenfalls zwei Substrate (bi) in zwei Produkte (bi) umgesetzt werden, allerdings in separaten Reaktionsschritten. So setzen Transaminasen ihr Substrat A (Aminosäure 1) zu einem Produkt P (alpha-Ketosäure 1) P um (Ping). Nachfolgend wird eine andere alpha-Ketosäure (B)aufgenommen und die analoge Aminosäure überführt (Pong). Dieser Mechanismus gelingt, da die prosthetische Gruppe des Enzyms (Pyridoxalphosphat) zwischen den Reaktionen zu Pyridoxaminphosphat wird. Man spricht vom Übergang der Enzymform „E“ in die Enzymform „F“. Am Ende des Zyklus ist der Ausgangszustand wiederhergestellt.

Abb. 2: Reaktionswege für Zweisubstrat-Reaktionen (zwei Prototypen)
- Sequenzieller bi-bi Mechanismus: Im Fall eines geordneten Ablaufes (´obligatory order´) ist die Bindung des ersten Substrates (A) notwendig, damit das Enzym die Bindungsstelle für das zweite Substrat (B) ausbilden kann, das heißt das „Leitsubstrat“ (NADH,H+) muss assoziiert sein, bevor das Folgesubstrat (Pyruvat) binden kann. Entsprechend muss das Leitprodukt P (NAD+) den EPQ-Komplex verlassen haben, bevor Q (Lactat) aus EQ abdissoziieren kann. Komplexe der Art EAQ und EBP (Substrat und Partnerprodukt) gibt es grundsätzlich nicht. Auch Komplexe wie EP bzw. EB haben beim geordnet-sequenziellen Mechanismus keine Existenzberechtigung. Im Gegensatz hierzu assoziieren/dissoziieren beim zufälligen (´random order´) Mechanismus alle Substrate/Produkte in beliebiger Folge. Komplexe des Typs EB und EP können existieren (nicht gezeigt).
- Ping-pong bi-bi Mechanismus. Ein Produkt wird bereits nach Bindung des ersten Substratmoleküls freigesetzt (Ping). E geht in eine andere Enzymform (F) über, mit der das zweite Substrat unter Bildung des zweiten Produktes reagiert (Pong).
Zur Unterscheidung beider Reaktionstypen kann eine Analyse wie in Abb. 1 dienen:
- die Umsatzgeschwindigkeit von Pyruvat (B) wird in Gegenwart verschiedener (nichtsättigender)Konzentrationen an NADH,H+ (A) untersucht. Nur diejenigen Enzymmoleküle, die bereits NADH,H+ gebunden haben, können auch Pyruvat (B) binden und die Reaktion ausführen. Die maximale Reaktionsgeschwindigkeit, Vmax, kann nicht erreicht werden. Diejenigen Enzymmoleküle allerdings, die als Komplex EAB vorliegen, ergeben für A und B die erwarteten Michaeliskonstanten (Km);
- im Falle eines Enzyms nach dem Ping-pong Mechanismus würde in Abb. 1A eine Schar paralleler Geraden erhalten (nicht gezeigt).
Da sequenziell arbeitende Enzyme verbreitet, Ping-Pong-Enzyme hingegen seltener sind, sollen hier nur die Eigenschaften der ersten Gruppe vertieft werden.
Sequenzielle Mechanismen: geordnet oder willkürlich?
Die drei Grundregeln:
- Anhand der LDH wurde die obligate Assoziation des „Leitsubstrates“ A (NADH,H+) vor dem „Folgesubstrat“ B (Pyruvat) beschrieben. Dies hat zur Folge, dass das Enzym als EA und EAB-Komplex vorliegen kann, nicht aber als Komplex EB (Regel 1).
- Analog hat das „Leitprodukt“ P (NAD+) das Enzym vor dem „Folgeprodukt“ Q (Lactat) zu verlassen, das heißt, ein Komplex EP kann nicht existieren, wohl aber ein Komplex EPQ bzw. EQ (Regel 2).
- Weiterhin gibt es keine „Mischformen“ EAQ und EBP, in denen jeweils ein Substrat und ein Produkt vereinigt wären (Regel 3).
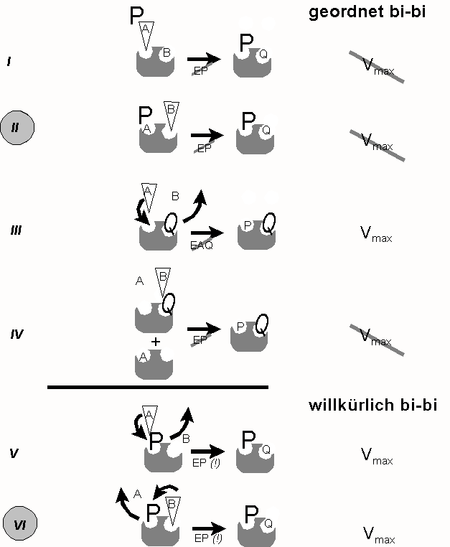
Führt man nun enzymkinetische Analysen analog zur Abb. 1, jedoch in Gegenwart eines großen Überschusses eines der Produkte (P oder Q) durch, so lässt sich auch der geordnete Ablauf beweisen und zwar dadurch dass die maximale Reaktionsgeschwindigkeit in Gegenwart des Produktes (NAD+) nicht erreicht wird (Abb. 3, Situation II). Beim willkürlichen Mechanismus würde sie hingegen erreicht (Abb. 3 Situation VI). Das nachfolgende Schema widmet sich den Variationsmöglichkeiten einer solchen Versuchsführung im Detail:
Abb. 3: Kinetiken in Gegenwart eines Produkt-Überschusses - Unterscheidung geordneter (´obligatory order´) und willkürlicher (´random order´) Reaktionsmechanismen- Großbuchstaben: Produkt im Überschuss
- Keil: Substrat, dessen Konzentration variiert wird
I: A und B treffen auf E; P kann sich nur mit EQ verbinden (Regel 2), was Konversion von B voraussetzt. Steigende [A] beschleunigt EPQ-Bildung; Vmax wird nicht erreicht, da EPQ-Dissoziation durch P behindert wird (Muster: „nichtkompetitiv“)
II: A und B treffen auf E. P kann sich nur mit EQ verbinden, was Konversion von B voraussetzt., Steigende [B] beschleunigt EPQ-Bildung; Vmax wird nicht erreicht, da EPQ-Dissoziation durch P behindert wird (Muster: „nichtkompetitiv“)
III: A und B treffen auf EQ. Da es keinen Komplex EAQ gibt (Regel 3), steht A in Kompetition zu Q: steigende [A] führen zur Verdrängung des störendenden Nachbarn wobei Vmax erreicht wird, Km aufgrund der Kompetition jedoch steigt (Muster: „kompetitiv“)
IV: A und B treffen auf EQ. Steigende [B] kann Q nicht verdrängen, da Bindung von B nur an EA erfolgen könnte (Regel 1). Vmax kann nicht erreicht werden, da Bindungsplatz für A stets nur teilgesättigt sein kann (Muster: „nichtkompetitiv“)
V: A und B treffen auf EP. Steigende [A] ist in der Lage, das im selben Bindungplatz befindliche Produkt zu verdrängen, so dass Vmax erreicht wird (Muster: "kompetitiv")
VI: A und B treffen auf EP. Steigende B verdrängt P aus Nachbarbindungsplatz, da EPB nicht existieren kann (Regel 3). Vmax wird erreicht, da Dissoziation von Q aus EPQ durch P nicht behindert wird (Muster: „kompetitiv“)
Weblinks
Literatur
- Cleland, W. W. (1963) The kinetics of enzyme-catalyzed reactions with two or more substrates. Biochem. Biophys. Acta 67, 104–137; 67, 173–187 und 67, 188–196.
- Bisswanger, H. (1994) Enzymkinetik – Theorie und Methoden (2. neu bearbeitete Auflage) Kapitel 2.6 "Mehrsubstratreaktionen" VCH Weinheim.
- Voet, D., Voet, J.G. und Pratt, C. (2002) Lehrbuch der Biochemie, Kap. 12, Enzymkinetik, Hemmung und Regulation, pp 353 Wiley VCH
Wikimedia Foundation.