- EGFP
-
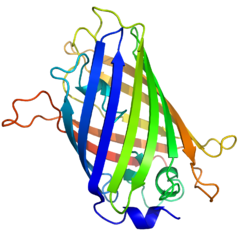
Grün fluoreszierendes Protein Bändermodell nach PDB 1EMA Vorhandene Strukturdaten: siehe UniProt-Eintrag Größe 238 Aminosäuren, 26,9 kDa Struktur Monomer oder Dimer Bezeichner Gen-Name(n) GFP Externe IDs UniProt: P42212 Vorkommen Übergeordnetes Taxon Aequorea victoria Das grün fluoreszierende Protein (Abkürzung GFP; engl. green fluorescent protein) ist ein erstmals 1961 von Osamu Shimomura[1][2] beschriebenes Protein aus der Qualle Aequorea victoria, das bei Anregung mit blauem oder ultraviolettem Licht grün fluoresziert. Seine unschätzbare Bedeutung in der Biologie, insbesondere der Zellbiologie, liegt in der Möglichkeit, GFP mit beliebigen anderen Proteinen Gen-spezifisch zu fusionieren. Durch die Fluoreszenz des GFP kann so die räumliche und zeitliche Verteilung des anderen Proteins in lebenden Zellen, Geweben oder Organismen direkt beobachtet werden.
Im Jahr 2008 wurde der Nobelpreis für Chemie für die „Entdeckung und Weiterentwicklung des grün fluoreszierenden Proteins“ an Osamu Shimomura, Martin Chalfie und Roger Tsien verliehen.
Inhaltsverzeichnis
Eigenschaften
Die Primärstruktur besteht aus 238 Aminosäuren mit einer Molekülmasse von 26,9 kDa.[3] Der eigentliche Fluorophor des GFP bildet sich offenbar autokatalytisch aus der Tripeptidsequenz Ser65–Tyr66–Gly67 innerhalb der Polypeptidkette. Diese intrinsische Fluoreszenz basiert nicht auf einem Umbau durch ein externes Enzym oder nachträglich integrierte Substanzen, kommt also vollständig ohne eventuell zellspezifische Prozessierungssysteme aus.
In seinem Ursprungsorganismus erhält GFP seine Anregungsenergie durch strahlungsfreien Energietransfer vom Photoprotein Aequorin. In Anwendungen wird GFP immer optisch angeregt. Das unmodifizierte, natürlich vorkommende GFP hat zwei Anregungsmaxima. Das erste liegt bei einer Wellenlänge von 395 nm, das zweite bei 475 nm. Die Emissionswellenlänge liegt bei 509 nm.
Anwendung
Douglas Prasher isolierte, klonierte und sequenzierte 1992 die cDNA von GFP.[4] Seit es Prasher 1994 gelang, GFP als Marker für andere Proteine zu benutzen, ist diese Technik in wenigen Jahren zu einer Standardmethode der Zellbiologie geworden.[5] Zur Herstellung von GFP-Fusionsproteinen wird die DNA des zu untersuchenden Proteins mit der GFP-DNA verbunden und in eine Form gebracht (siehe Vektor), die von der Zelle aufgenommen werden kann, so dass sie das Fusionsprotein selbstständig herstellt (Transfektion/oder Transformation bei nicht Zellkulturen). In vielen Fällen wird das zu untersuchende Protein noch an die korrekte Stelle in der Zelle transportiert, und das GFP kann durch Fluoreszenzmikroskopie Aufschluss über die zeitliche und räumliche Lokalisation des Zielproteins in der Zelle geben. Bei der Photoactivated Localization Microscopy werden durch Licht aktivierbare fluoreszierende Proteine genutzt, um die lichtmikroskopische Auflösung zu verbessern.
GFP ist in nahezu allen eukaryontischen Zellen als nicht-toxisch einzustufen und eignet sich daher perfekt für die Untersuchung biologischer Prozesse in vivo. Einziges Problem kann bei sehr hoher Expression die Bildung von Peroxid bei der Entstehung des Fluorophors sein, welches die Zelle unter Stress setzen und schädigen könnte.
Auch kuriose Anwendungen, wie z. B. ein Leuchtkaninchen oder ein in den USA im Tierhandel unter dem Namen GloFish erhältlicher, genetisch manipulierter Zebrabärbling (Danio rerio) sind zu finden.[6]
Varianten
Mittlerweile gibt es diverse modifizierte Versionen des Original-GFP, die andere Fluoreszenzspektren aufweisen. Entsprechend der Farbe heißen diese zum Beispiel CFP (cyan) oder YFP (yellow). Bei geschickter Anwendung sind einzelne Zellorganellen unterschiedlich einfärbbar und mittels Entmischung (spektrale Dekonvolution) dann getrennt beobachtbar. Als entscheidend ist auch noch die Entwicklung von enhanced-Varianten wie z. B. dem enhanced GFP (eGFP) oder enhanced YFP (eYFP) zu sehen.
Immer größere Bedeutung bekommen auch fluoreszente Proteine aus Korallen (Anthozoa).[7] Zu nennen sind hier die zoanFP (aus Zoanthus sp.) oder auch das rot fluoreszierende Protein drFP583 (aus Discosoma), Handelsname DsRed. Viele dieser Proteine wurden bereits mutiert und in ihren Eigenschaften verändert um andere Eigenschaften zu gewinnen. Viele fluoreszierende Proteine sind Tetramere. Dies wird genutzt, indem man sich unterschiedlich schnell entwickelnde Monomere einbaut, dabei verändert sich also die Farbe des Proteins mit der Zeit.[8] Solche Fluoreszenz-Timer sind extrem nützlich, um beispielsweise das Alter von Organellen feststellen zu können. Die DsRed-Mutante E5 hat beispielsweise diese Eigenschaft.
Quellen
- ↑ Osamu Shimomura, F. H. Johnson und Y. Saiga: Extraction, purification and properties of aequorin, a bioluminescent protein from the luminous hydromedusan, Aequorea. In: Journal of Cellular and Comparative Physiology. Band 59, 1962, S. 223-239. PMID 13911999 doi:10.1002/jcp.1030590302
- ↑ Osamu Shimomura: The discovery of aequorin and green fluorescent protein. In: Journal of Microscopy. Band 217, 2005, S. 1-15. PMID 15655058 doi:10.1111/j.0022-2720.2005.01441.x PDF
- ↑ UniProt P42212
- ↑ D. C. Prasher, V. K. Eckenrode, W. W. Ward, F. G. Prendergast und M. J. Cormier: Primary structure of the Aequorea victoria green-fluorescent protein. In: Gene. Band 111, 1992, S. 229-233. PMID 1347277
- ↑ M. Chalfie, Y. Tu, G. Euskirchen, W.W. Ward und D.C. Prasher: Green fluorescent protein as a marker for gene expression. In: Science. Band 263, 1994, S. 802-805. PMID 8303295 doi:10.1126/science.8303295
- ↑ Transgene Aquariumsfische. Telepolis, 23.11.2003.
- ↑ V.V. Verkusha und K.A. Lukyanov: The molecular properties and applications of Anthozoa fluorescent proteins and chromoproteins. In: Nature Biotechnology. Band 22, 2004, S. 289-296. PMID 14990950 doi:10.1038/nbt943
- ↑ A. Terskikh et al.: "Fluorescent Timer". Protein That Changes Color With Time. In: Science. Band 290, 2000, S. 1585-1588. PMID 11090358 doi:10.1126/science.290.5496.1585
Literatur
- R.Y. Tsien: The green fluorescent protein. In: Annual Reviews in Biochemistry. Band 67, 1998, S. 509-544. PMID 9759496 doi:10.1146/annurev.biochem.67.1.509
- Martin Chalfie und Steven Kain: GFP: Properties, Applications and Protocols. 2. Auflage, Wiley & Sons, Hoboken 2005, ISBN 0471736821
- M. Zimmer: Glowing Genes. A Revolution in Biotechnology. Prometheus Books, Buffalo, NY 2005, ISBN 1591022533
- Daniel Veith, Martina Veith: Biologie fluoreszierender Proteine: Ein Regenbogen aus dem Ozean Biologie in unserer Zeit, 6/2005, S. 394-404. doi:10.1002/biuz.200410295
Siehe auch
- GFP-cDNA
- Moderne Untersuchungsmethoden in der Zellbiologie: FRET
- Leuchtkaninchen (Anwendung in der Kunst)
Weblinks
- Green Fluorescent Protein – History (engl.)
- Molecule of the Month – Green Fluorescent Protein (GFP) (engl.)
- Interpro: Protein Of The Month: Green Fluorescent Protein. (engl.)
- Umweltforschungszentrum: Mikrobiologen bringen Bakterien zum Leuchten.
- The Nobel Prize in Chemistry 2008 (engl.)
- Proteopedia: Green Fluorescent Protein (engl.)
Wikimedia Foundation.