- Foldit
-
Foldit 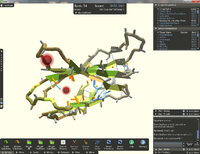
Screenshot von Foldit während ein Puzzle geladen wurdeBasisdaten Entwickler University of Washington
Departments of Computer Science & Engineering and BiochemistryBetriebssystem Windows, Mac OS X, Linux Programmiersprache C++ Lizenz Proprietär http://fold.it/ Foldit ist ein experimentelles Videospiel, das in Zusammenarbeit der Abteilungen „Computer Science and Engineering“ und „Biochemistry“ der University of Washington entstanden ist. Beteiligt sind viele Leute des Rosetta@home-Projektes. Die erste öffentliche Beta wurde im Mai 2008 veröffentlicht.
Ziel des Spieles ist es, ein möglichst gut „gefaltetes“ Protein zu erhalten, d.h. ein Modell des Proteins im Zustand des Energieminimums. Das ist die Form, in der es in der Natur vorkommt. Dazu sind allerdings keinerlei Vorkenntnisse nötig, die Bewertung erledigt das Programm. Die Möglichkeiten, die der Spieler zur Proteinmanipulation hat, werden in einer Serie von Tutorialpuzzles erklärt. Für das Spiel wird dabei eine graphische Entsprechung der Proteinstruktur angezeigt, die der Spieler mit verschiedenen Werkzeugen verändern kann.
Wenn die Struktur verändert wird, berechnet das Programm einen Punktwert basierend darauf, wie gut das Protein gefaltet ist. Für jedes Puzzle wird eine High Score sowohl für Einzel- als auch für Gruppenlösungen erstellt, die sich in Echtzeit ändert.
Der Prozess, mit dem Lebewesen die primäre Struktur eines Proteins synthetisieren, die Proteinbiosynthese, ist recht gut verstanden, ebenso die Kodierung als DNA. Zu bestimmen, wie die primäre Struktur eines Proteins sich in eine funktionierende, dreidimensionale Struktur verwandelt – wie sich das Protein faltet – ist schwieriger. Der generelle Prozess ist bekannt, aber die Vorhersage von Proteinstrukturen verlangt viel Rechenzeit, stark steigend mit zunehmender Länge des Proteins, und ist unvollkommen.
Foldit ist der Versuch, die natürlichen menschlichen 3-D-Mustererkennungsfähigkeiten auf dieses Problem anzusetzen. Gegenwärtige Puzzles basieren auf gut verstandenen Proteinen. Indem untersucht wird, auf welche Art die Spieler intuitiv an diese Puzzle herangehen, versuchen die Wissenschaftler, existierende Proteinfaltungssoftware zu verbessern. 2008 nahm Foldit am CASP 2008 Protein-Vorhersage-Wettbewerb teil, und schnitt trotz geringer Erfahrung der Spieler und teils unausgereifter Werkzeuge sehr gut ab. In der Hälfte der Fälle gelang eine Top-3-Platzierung und einmal der Spitzenplatz (bei 71 bis 83 teilnehmenden Laboren, zwei Mal 527). In jedem einzelnen Fall wurden alle Modelle, die nur von Computern erstellt wurden, übertroffen.
Zu den Erfolgen von Foldit zählt unter anderem die Entschlüsselung der Struktur des monomerischen Proteins M-PMV-Protease (Mason-Pfizer monkey virus retroviral protease), das AIDS bei Rhesusaffen auslöst.[1][2][3]
Foldit ist ein Mix aus Crowdsourcing und Verteiltem Rechnen.
Literatur
- S. Cooper, F. Khatib, A. Treuille, J. Barbero, J. Lee, M. Beenen, A. Leaver-Fay, D. Baker, Z. Popović, F. Players: Predicting protein structures with a multiplayer online game. In: Nature. Band 466, Nummer 7307, August 2010, S. 756–760, ISSN 1476-4687. doi:10.1038/nature09304. PMID 20686574. PMC 2956414.
- F. Khatib, F. Dimaio, S. Cooper, M. Kazmierczyk, M. Gilski, S. Krzywda, H. Zabranska, I. Pichova, J. Thompson, Z. Popović, M. Jaskolski, D. Baker: Crystal structure of a monomeric retroviral protease solved by protein folding game players. In: Nature structural & molecular biology. September 2011, ISSN 1545-9985. doi:10.1038/nsmb.2119. PMID 21926992.
- Firas Khatib, Seth Cooper, Michael D. Tyka, Kefan Xu, Ilya Makedon, Zoran Popović, David Baker, and Foldit Players: Algorithm discovery by protein folding game players. In: Proceedings of the National Academy of Sciences of the United States of America. November 2011, ISSN 1091-6490. doi:10.1073/pnas.1115898108.
Einzelnachweise
- ↑ Gamer klären Struktur eines Virus-Enzyms auf. Spiegel Online, 19. September 2011.
- ↑ Alan Boyle: Gamers solve molecular puzzle that baffled scientists. Cosmic Log, 18. September 2011.
- ↑ F. Khatib, F. Dimaio, S. Cooper, M. Kazmierczyk, M. Gilski, S. Krzywda, H. Zabranska, I. Pichova, J. Thompson, Z. Popović, M. Jaskolski, D. Baker: Crystal structure of a monomeric retroviral protease solved by protein folding game players. In: Nature structural & molecular biology. ISSN 1545-9985. doi:10.1038/nsmb.2119. PMID 21926992.
Weblinks
- Foldit Projektseite
- Foldit Wiki (englisch, „offizielles“ Wiki)
- Deutsches Wiki
- NZZ: Proteine falten als Online-Zeitvertreib (Zugriff am 29-Sep-2011)
Wikimedia Foundation.
