- Proteinstruktur
-
Die Proteinstruktur oder Konformation wird in der Biochemie in verschiedene Strukturebenen eingeteilt. Die Einteilung zu einer Hierarchie in Primärstruktur, Sekundärstruktur, Tertiärstruktur und Quartärstruktur wurde erstmals 1952 von Kaj Ulrik Linderstrøm-Lang vorgeschlagen.[1]
Inhaltsverzeichnis
Die Hierarchie der Strukturebenen
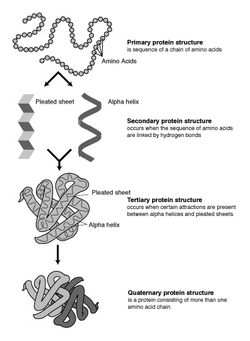
In der Biochemie werden vier hierarchisch angeordnete Strukturebenen in Proteinen unterschieden:
- Primärstruktur – die Aminosäuresequenz der Peptidkette.
- Sekundärstruktur – die räumliche Struktur eines lokalen Bereiches im Protein (z.B. α-Helix, β-Faltblatt).
- Tertiärstruktur – die räumliche Struktur des einzelnen Proteins bzw. einer Untereinheit.
- Quartärstruktur – die räumliche Struktur des gesamten Proteinkomplexes mit allen Untereinheiten.
Einige Proteine ordnen sich noch in einer über die Quartärstruktur hinausgehenden, molekular aber bereits ebenso prädeterminierten "Überstruktur" oder "Suprastruktur" an, wie Kollagen in der Kollagenfibrille oder Aktin, Myosin und Titin im Sarkomer der Muskelfibrille oder Kapsomeren im Kapsid behüllter Viren.
Strukturbestimmung
Zur Aufklärung der räumlichen Proteinstruktur stehen verschiedene experimentelle Methoden zur Verfügung:
- Bei der Kristallstrukturanalyse wird - meist mittels Röntgenstrahlen ein Beugungsbild eines Proteinkristalls erstellt, aus dem sich dann die dreidimensionale Struktur errechnen lässt. Es ist allerdings recht kompliziert, die hierfür benötigten Einkristalle herzustellen; für manche Proteine ist es bisher unmöglich. Ein weiteres Problem bei diesem Verfahren besteht darin, dass die Struktur der Proteine im Kristall nicht unbedingt der natürlichen Struktur entspricht, da sich z.B. durch die Kristallisation neue Kontakte bilden, die sonst nicht vorhanden wären (crystal packing). Für auswertbare Beugungsbilder ist eine Mindestgröße der Proteinkristalle erforderlich. Um die erforderliche Substanzmenge zu erhalten, wird oft auf von Bakterien hergestellte Proteine zurückgegriffen, welche mitunter nicht die in höheren Organismen vorhandenen posttranslationalen Modifikationen aufweisen.
- Mittels NMR (NMR von engl. nuclear magnetic resonance) kann die Struktur eines Proteins in Lösung ermittelt werden, was näher an den physiologischen ("natürlichen") Bedingungen ist. Hierbei ergibt sich keine eindeutige Struktur, da sich die Atome des Proteins in diesem Zustand bewegen. Um eine "eindeutige" Struktur zu erhalten, wird meist über die abgebildeten Strukturen gemittelt. Auch NMR kann bisher nicht für alle Proteinarten durchgeführt werden. Insbesondere die Größe ist hier ein begrenzender Faktor, denn Proteine > 30 kDa können bisher nicht analysiert werden, da die NMR-Ergebnisse zu komplex sind und man daraus keine eindeutige Proteinstruktur mehr ablesen kann.
Strukturvorhersage
Die Vorhersage von räumlichen Proteinstrukturen erzielt gute Ergebnisse, wenn es bereits Proteine mit ähnlicher Sequenz und bekannter Struktur gibt. Dann kann man das sogenannte homology modelling verwenden, wobei die neue Sequenz auf die alte abgebildet und damit in die Struktur "eingepasst" wird. Diese Technik ähnelt dem Sequenzalignment.
Schwieriger ist die Vorhersage, wenn noch keine ähnlichen Strukturen bekannt sind. Das Levinthal-Paradox zeigt, dass es viel zu viele mögliche Konformationen gibt, als dass man sie alle berechnen und die günstigste auswählen könnte. In der Bioinformatik sind zwar verschiedene Methoden der de novo- oder ab initio-Strukturvorhersage entwickelt worden, aber obwohl in den letzten Jahren große Fortschritte gemacht wurden, gibt es bisher keine wirklich zuverlässige Methode.
Um neue Methoden zur Strukturvorhersage miteinander vergleichen zu können, gibt es seit einigen Jahren den CASP-Wettbewerb (critical assessment of techniques for protein structure prediction). In diesem Wettbewerb werden Aminosäuresequenzen von Strukturen, an denen Kristallographen gerade arbeiten, für die Teilnehmer zur Verfügung gestellt. Die Teilnehmer verwenden ihre eigenen Methoden, um die Strukturen vorherzusagen. Ein Auswertungs-Team vergleicht die Vorhersagen dann mit den experimentell ermittelten Strukturen.
Die Strukturvorhersage ist auch Ziel mehrerer Projekte des verteilten Rechnens wie z.B. Rosetta@home, POEM@home, Predictor@home, Folding@home und Human Proteome Folding Project. Das Spiel Foldit macht sich zudem zur Strukturaufklärung die Vorteile des Crowdsourcing zu nutze.
Quellen
- ↑ Linderstrøm-Lang, K.U. (1952): Proteins and Enzymes. In: Lane Medical Lectures. Bd. 6, S. 1-115. Stanford University Publications, University Series, Medical Sciences, Stanford University Press.
Weblinks
- Dr. Ulrich Marsch (TU München): Bioinformatik-Strategie erfolgreich bei der Strukturaufklärung von Membranproteinen. Pressemitteilung. In: idw-online. Informationsdienst Wissenschaft e. V., 28. Oktober 2010, abgerufen am 29. Oktober 2010.
Wikimedia Foundation.