- HMM
-
Das Hidden Markov Model (HMM) ist ein stochastisches Modell, das sich durch zwei Zufallsprozesse beschreiben lässt. Ein Hidden-Markov-Model ist auch die einfachste Form eines dynamischen Bayesschen Netz.
Der erste Zufallsprozess entspricht dabei einer Markow-Kette, die durch Zustände und Übergangswahrscheinlichkeiten gekennzeichnet ist. Die Zustände der Kette sind von außen jedoch nicht direkt sichtbar (sie sind hidden, verborgen). Stattdessen erzeugt ein zweiter Zufallsprozess zu jedem Zeitpunkt beobachtbare Ausgangssymbole (oder Beobachtungen) gemäß einer zustandsabhängigen Wahrscheinlichkeitsverteilung. Das Attribut versteckt bezieht sich bei einem Hidden Markov Model auf die Zustände der Markow-Kette während der Ausführung, nicht auf die Parameter des Markow-Modells. Die Aufgabe besteht häufig darin, aus der Sequenz der Ausgabesymbole (Beobachtungen) auf die Sequenz der verborgenen Zustände zu schließen.
Wichtige Anwendungsgebiete sind neben der Spracherkennung (und allgemein Computerlinguistik) und der Bioinformatik unter Anderem Spamfilter (insbesondere Markow-Filter), Gestenerkennung in der Mensch-Maschine-Kommunikation (Robotik), Schrifterkennung und Psychologie.
Inhaltsverzeichnis
Definition
Formal definiert man ein HMM als Quintupel λ = (S,A,B,π,V) mit
- S = {s1,...,sn} – Menge aller Zustände
- A = {aij} – Zustandsübergangsmatrix, wobei aij die Wahrscheinlichkeit angibt, dass von Zustand si in Zustand sj gewechselt wird.
- B = {b1,...,bn} – Menge der Emissionswahrscheinlichkeitsverteilungen bzw. Dichten
- π – Anfangswahrscheinlichkeitsverteilung mit π(i) Wahrscheinlichkeit, dass si der Startzustand ist
- V – Beobachtungsmatrix wobei bsi die Wahrscheinlichkeit angibt, im Zustand s die Beobachtung
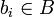 zu machen
zu machen
Ein HMM ist stationär wenn sich die Übergangs- und Emmisionswahrscheinlichkeiten nicht mit der Zeit ändern. Diese Annahme ist oft sinnvoll, weil auch die modellierten Naturgesetze konstant sind. Im Normalfall sind die unterliegenden Markow-Ketten erster Ordnung, das heißt der aktuelle Zuständ hängt nur von genau einem vorherigen Zustand ab.
Veranschaulichung
Das Bild zeigt die generelle Architektur eines instantierten HMMs. Jedes Oval ist die Repräsentation einer zufälligen Variable, welche beliebige Werte aus S annehmen kann. Die Zufallsvariable x(t) ist der versteckte Zustand des HMMs zum Zeitpunkt t. Die Zufallsvariable y(t) ist die Beobachtung zum Zeitpunkt t. Die Pfeile in dem Trellis Diagramm bedeuten eine bedingte Abhängigkeit.
Im obigen Diagramm sieht man, dass der Zustand der versteckten Variable x(t) zum Zeitpunkt t nur vom Zustand der Variable x(t − 1) abhängt, frühere Werte haben keinen weiteren Einfluss. Deshalb ist das Modell ein Markov-Modell 1. Ordnung. Wenn höhere Ordnungen gebraucht werden, können diese durch das Einfügen neuer versteckter Zustände auf die 1. Ordnung zurückgeführt werden. Der Wert von y(t) hängt von x(t) ab.
Problemstellungen
Im Zusammenhang mit HMMs existieren fünf grundlegende Problemstellungen.
Filtern
Gegeben ist ein HMM λ sowie eine Beobachtungssequenz Y1:t. Gesucht ist die Wahrscheinlichkeit P(Xt | Y1:t), d.h. der momentane verborgene Zustand X zum Zeitpunkt t. Ein effizientes Verfahren zur Lösung des Evaluierungsproblems ist der Forward-Algorithmus.
Prediktion
Gegeben ist ein HMM λ und die Beobachtungssequenz Y1:t. Gesucht ist Wahrscheinlichkeit P(Xt + δ | Y1:t), also die Wahrscheinlichkeit, dass sich das HMM zum Zeitpunkt t + δ im Zustand X befindet unter der Bedingung, dass die Ausgabe Y beobachtet wurde. Prediktion ist quasi wiederholtes Filtern ohne neue Beobachtungen und lässt sich auch einfach mit dem Forward-Algorithmus berechnen.
Glätten
Gegeben ist ein HMMλ und die Beobachtungssequenz Y1:t Gesucht ist die Wahrscheinlichkeit P(Xt − δ | Y1:t), also die Wahrscheinlichkeit, dass sich das Model zu einem früheren Zeitpunkt im Zustand X befand, unter der Bedingung, dass Y beobachtet wurde. Mit Hilfe des Backward-Algorithmus kann diese Wahrscheinlichkeit effizient berechnet werden.
Dekodierungsproblem
Gegeben ist ein HMM λ und die Beobachtungssequenz Y1:t. Es soll die wahrscheinlichste Sequenz der (versteckten, hidden) Zustände bestimmt werden, die eine vorgegebene Ausgabesequenz erzeugt hat. Lösbar mit Hilfe des Viterbi-Algorithmus.
Lernproblem
Gegeben ist nur die Ausgabesequenz. Es sollen die Parameter des HMM bestimmt werden, die am wahrscheinlichsten die Ausgabesequenz erzeugen. Lösbar mit Hilfe des Baum-Welch-Algorithmus.
Beispiel
Ein Gefangener im Kerkerverlies möchte das aktuelle Wetter herausfinden. Er weiß, dass auf einen sonnigen Tag zu 70 % ein Regentag folgt und dass auf einen Regentag zu 50 % ein Sonnentag folgt. Weiß er zusätzlich, dass die Schuhe der Wärter bei Regen zu 90 % dreckig, bei sonnigem Wetter aber nur zu 60 % dreckig sind, so kann er durch Beobachtung der Wärterschuhe Rückschlüsse über das Wetter ziehen.
Zur Untersuchung von DNA-Sequenzen mit bioinformatischen Methoden kann das HMM verwendet werden. Beispielsweise lassen sich so CpG-Inseln in einer DNA-Sequenz aufspüren. Dabei stellt die DNA-Sequenz die Beobachtung dar, deren Zeichen {A,C,G,T} bilden das Ausgabealphabet. Im einfachsten Fall besitzt das HMM zwei verborgene Zustände, nämlich CpG-Insel und nicht-CpG-Insel. Diese beiden Zustände unterscheiden sich in ihrer Ausgabeverteilung, so dass zum Zustand CpG-Insel mit größerer Wahrscheinlichkeit Zeichen C und G ausgegeben werden.
Anwendungsgebiete
Anwendung finden HMMs häufig in der Mustererkennung bei der Verarbeitung von sequentiellen Daten, z. B. physikalische Messreihen, aufgenommene Sprachsignale oder biologische Sequenzen. Dazu werden die Modelle so konstruiert, dass die verborgenen Zustände semantischen Einheiten entsprechen (z. B. Phoneme in der Spracherkennung), die es in den sequentiellen Daten (z. B. Kurzzeit-Spektren des Sprachsignals) zu erkennen gilt. Eine weitere Anwendung besteht darin, für ein gegebenes HMM durch eine Suche in einer Stichprobe von sequentiellen Daten solche Sequenzen zu finden, die sehr wahrscheinlich von diesem HMM erzeugt sein könnten. Beispielsweise kann ein HMM, das mit Vertretern einer Proteinfamilie trainiert wurde, eingesetzt werden, um weitere Vertreter dieser Familie in großen Proteindatenbanken zu finden.
Geschichte
Hidden Markov Modelle wurden erstmals von Leonard E. Baum und anderen Autoren in der zweiten Hälfte der 1960er Jahre publiziert. Eine der ersten Applikationen war ab Mitte der 1970er die Spracherkennung. Seit Mitte der 1980er wurden HMMs für die Analyse von DNA-Sequenzen eingesetzt und sind seitdem fester Bestandteil der Bioinformatik.
Literatur
- Lawrence R. Rabiner: A Tutorial on Hidden Markov Models and Selected Applications in Speech Recognition. Proceedings of the IEEE, Band 77, Nr. 2, S. 257–286, 1989 (PDF; 2,2 MB).
- Rainer Merkl, Stephan Waack: Bioinformatik interaktiv. Wiley-VCH, 2002, ISBN 3-527-30662-5.
- Gernot A. Fink: Mustererkennung mit Markov-Modellen: Theorie, Praxis, Anwendungsgebiete. Teubner, 2003, ISBN 3-519-00453-4
Weblinks
- http://code.google.com/p/jahmm/ HMM Java-Bibliothek, die unter der neuen BSD-Lizenz verfügbar ist.
- http://www.ghmm.org HMM C-Bibliothek, die unter der LGPL frei verfügbar ist
- Seminarvortrag zum Thema: Hidden Markov Prozesse und das Suchen von Sequenzen
Wikimedia Foundation.