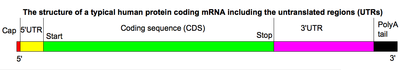
- MRNA
-
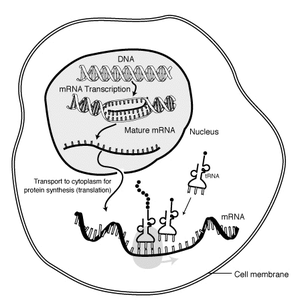 Die Funktion der mRNA in einer eukaryotischen Zelle. DNA wird im Zellkern mittels RNA transkribiert. Die prozessierte mRNA wird in das Cytoplasma transportiert und an den Ribosomen für die Proteinsynthese translatiert.
Die Funktion der mRNA in einer eukaryotischen Zelle. DNA wird im Zellkern mittels RNA transkribiert. Die prozessierte mRNA wird in das Cytoplasma transportiert und an den Ribosomen für die Proteinsynthese translatiert.Als mRNA (von engl. messenger RNA), auch Boten-RNA, wird das RNA-Transkript eines zu einem Gen gehörigen Teilabschnitts der DNA bezeichnet. Die mRNA wird bei der Transkription von dem Enzym RNA-Polymerase synthetisiert.
Transkription
Die Transkription geschieht bei Prokaryoten im Cytoplasma, bei Eukaryoten im Zellkern (Karyoplasma). Der Prozess ist bei Prokaryoten (Lebewesen ohne Zellkern) und Eukaryoten (Lebewesen mit Zellkern) ähnlich, allerdings kann in Prokaryoten sofort die Translation (der eigentliche Vorgang der Synthese von Proteinen) beginnen. Ribosomen setzen sich an die mRNA, noch bevor die Transkription abgeschlossen ist. Die mRNA von Eukaryoten wird dagegen meist noch vor Ende der Transkription prozessiert und dann aus dem Zellkern ausgeschleust.
Prokaryoten besitzen im Gegensatz zu den Eukaryoten nur eine Art des Enzyms RNA-Polymerase, welches die Synthese von RNA übernimmt. Dagegen besitzen Eukaryoten drei RNA-Polymerasen:
Eukaryotische RNA-Polymerasen Enzym Produkt RNA-Polymerase I rRNA RNA-Polymerase II mRNA RNA-Polymerase III tRNA Ein weiterer wesentlicher Unterschied zwischen prokaryotischer und eukaryotischer mRNA besteht darin, dass prokaryotische mRNA polycistronisch sein kann, eukaryotische mRNA dagegen immer monocistronisch ist. Das ermöglicht es Prokaryoten auf nur einem einzigen mRNA-Transkript die Informationen von mehreren (auf der DNA hintereinanderliegenden) Genen zu haben. Ein solcher gemeinsam transkribierter Bereich heißt Operon. Auf diese Weise ist es möglich, dass bei Prokaryoten noch während der mRNA-Synthese bereits die ersten Proteine fertig translatiert werden.
Eukaryotische prä-mRNA-Prozessierung
In Eukaryoten muss die mRNA, die zunächst in einer Vorstufe (prä-mRNA oder hnRNA (von engl. heteronucleic RNA oder pre-mRNA)) vorliegt, erst prozessiert werden und dann durch die Poren des Zellkerns ins Cytoplasma gelangen, um dort von den Ribosomen erkannt zu werden, die mit der Proteinsynthese beginnen:
- Das 5'-Ende (dieses Ende wurde bei der Transkription zuerst synthetisiert) bekommt eine 5'-Cap-Struktur (vom englischen cap = Kappe). Diese Kappe besteht aus einer modifizierten Form des Guanosins. Die Cap-Struktur hat mindestens zwei wichtige Funktionen: Erstens wirkt sie nach dem Transport ins Cytosol als Signal für die kleine ribosomale Untereinheit, an die die mRNA andockt, und zweitens schützt sie die mRNA vor hydrolysierenden Enzymen.
- Das 3'-Ende, das bei der Transkription zum Schluss transkribiert wurde, erfährt eine Polyadenylierung. Bei diesem Vorgang wird ein Poly(A)-Schwanz aus 30 bis 200 Adenin-Nukleotiden angehängt, der die mRNA vor einem enzymatischen Abbau schützt. Zusätzlich erleichtert er den Export der mRNA aus dem Zellkern ins Cytoplasma.
- Durch Splicing werden die Introns, die keine kodierende Informationen enthalten, entfernt und die verbleibenden Exons miteinander verbunden. Dieser Vorgang findet im Spleißosom statt: Ein Komplex aus snRNPs (engl. Small Nuclear Ribonucleoproteins), bestehend aus den snRNAs U1, U2, U4, U5 und U6 und etwa 50 Proteinen, weiteren Spleißfaktoren und hnRNA. Durch alternatives Splicing können aus derselben prä-mRNA unterschiedliche mRNAs entstehen, die in der Translation zu unterschiedlichen Proteinen führen.
An dieser Stelle greifen auch diverse Regulationsprozesse der Zelle ein. Über Antisense-RNA und RNA-Interferenz kann mRNA wieder abgebaut werden und so die Translation verhindert werden. Weiterhin erfolgt in einigen Fällen die so genannte RNA-Edition, d. h. die Nukleotide in einer mRNA werden verändert. Ein Beispiel dafür ist die mRNA des Apolipoprotein B, die in manchen Geweben editiert wird, in anderen nicht. Durch die Edition entsteht ein früheres Stoppcodon, das bei der Translation zu einem kürzeren Protein (mit einer anderer Funktion) führt.
Translation und Degradation
Der Prozess der Translation (lat.: „Übertragung“, engl.: „Übersetzung“) findet an den Ribosomen statt, die sich (frei schwebend) im Cytoplasma oder angeheftet am endoplasmatischen Retikulum befinden.
Jedes Ribosom, das an die mRNA „anlegt“, „liest“ die mRNA, interpretiert sie anhand des „universellen genetischen Codes“ und synthetisiert ein entsprechendes Protein aus den in der Zelle verfügbaren Aminosäuren. Dieser Vorgang erzeugt in der Regel ein einziges Protein-Molekül. Danach löst sich das Ribosom von der mRNA und ein (anderes) Ribosom kann anlegen, wenn sie nicht vorher abgebaut wird. In der Regel lagern sich mehrere Ribosomen an eine mRNA an und synthetisieren das in ihr codierte Protein. Dieser Prozess wird auf natürliche Weise beschränkt, weil die benötigten Aminosäuren in einer Zelle irgendwann zur Neige gehen.
Irgendwann wird die mRNA durch das Enzym RNAse (Ribonuklease) wieder in ihre Einzelteile zerlegt, die dann von der Zelle wieder zum Aufbau neuer RNA-Moleküle genutzt werden können. Der Abbau-Prozess findet in den so genannten „P-Bodies“ statt, spezifischen Zellstrukturen im Cytoplasma. Diese erst kürzlich entdeckten Strukturen bieten eine weitere Form der Expressionsregulation, bei der die mRNA auch - statt abgebaut zu werden - für eine erneute Translation zwischengelagert werden kann.
Wikimedia Foundation.