- Influenzavirus
-
Influenzavirus  Influenzavirus A/Hong Kong/1/68 bei 70.000-facher Vergrößerung
Influenzavirus A/Hong Kong/1/68 bei 70.000-facher Vergrößerung
Systematik Reich: Viren Ordnung: nicht klassifiziert Familie: Orthomyxoviridae Taxonomische Merkmale Genom: (-)ssRNA linear, segmentiert Baltimore: Gruppe 5 Symmetrie: helikal Hülle: vorhanden Wissenschaftlicher Name Influenzavirus (engl.) Links NCBI Taxonomie: 197911 ICTVdB Virus Code: 00.046.0.01 Die drei Gattungen Influenzavirus innerhalb der Familie Orthomyxoviridae sind umhüllte Viren mit einer einzelsträngigen, segmentierten RNA von negativer Polarität als Genom. Unter den Gattungen finden sich auch die Erreger der Influenza oder „echten“ Grippe. Zu medizinischen Aspekten der Influenzaviren und Grippe-Erkrankung siehe Influenza.
Inhaltsverzeichnis
Aufbau
Virion
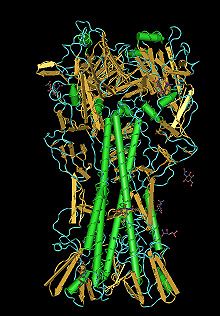
Das virale Nukleokapsid des Virions, ein Komplex aus dem Genom des Virus und bestimmten Proteinen, besitzt eine helikale Symmetrie.
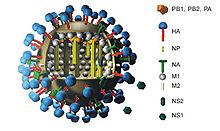
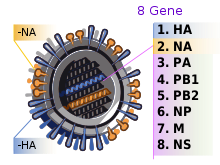
Im Elektronenmikroskop sieht man alle Gattungen dieses Virus als kugelige oder auch vielgestaltige, umhüllte Viruspartikel mit einem Durchmesser von 80 bis 120 nm, in deren Hülle eine jeweils unterschiedliche Anzahl an Proteinen und Glykoproteinen eingelagert sind. Diese Glykoproteine ragen als 10 bis 14 nm lange Spikes oder Peplomere genannte Fortsätze über die Virusoberfläche hinaus. Bei den Influenza-A- und Influenza-B-Viren sind genau zwei Typen dieser Spikes für die Virologen von besonderem Interesse: das Hämagglutinin (HA) und die Neuraminidase (NA).
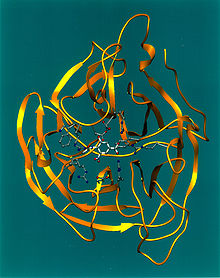
Das Hämagglutinin bewirkt die Verklumpung (Agglutination) von Erythrozyten und vermittelt bei der Infektion einer Wirtszelle die Anheftung und das Eindringen des Virus beziehungsweise des Virions. Dieses Ankoppeln geschieht durch eine Anlagerung eines kleinen Bereiches des Hämagglutininmoleküls an spezielle Proteine, Sialinsäure (SA), der Wirtszellenhülle, die als Rezeptoren (SA-Rezeptoren) fungieren.[1] Jede Hämagglutininvariante passt dabei (wie ein Schlüssel) in einen andersartigen besonderen Wirtszellenrezeptor (in ein Schloss), wobei jeder Wirt nur über einen Teil aller möglichen Rezeptoren verfügt. Diese Tatsache ist auch der Grund dafür, dass bestimmte Subtypen oder Virusvarianten mit ihrem speziellen Hämagglutinintyp bestimmte Wirte leicht infizieren und dabei eine Erkrankung auslösen können und andere prinzipiell mögliche Wirte wiederum nicht oder nur sehr eingeschränkt. Durch Virusmutationen besonders in Hinblick auf mögliche Veränderungen des Hämagglutinins kann sich die Infektionsgefahr für den einen oder anderen potentiellen Wirt erheblich ändern. Allerdings können die Viren die HA-Bindungsstelle nicht beliebig verändern, da diese auch eine wichtige Funktion in ihrem Vermehrungszyklus besitzt.[1]
Die Neuraminidase hat im Infektionsvorgang eine noch nicht abschließend geklärte enzymatische Funktion. Nach dem derzeitigen Erkenntnisstand spielt die Neuraminidase durch Zerstörung der N-Acetylneuraminsäure zellulärer Rezeptoren eine entscheidende Rolle bei der Freisetzung von durch die Replikation neu entstandenen Viren beziehungsweise Tochtervirionen aus den infizierten Zellen und damit einem Ausbreiten der Infektion sowohl innerhalb desselben Organismus als auch ein Übergreifen der Infektion auf andere Organismen. Außerdem verhindert die Neuraminidase das Hämagglutinin-vermittelte Anheften der Tochtervirionen an bereits infizierte Zellen. Zugleich verhindert sie, dass in einer infizierten Zelle ein Zelltodprogramm (Apoptose) gestartet werden kann, drosselt eine Bildung des Immunsystembotenstoffs Interferon und behindert damit eine Immunantwort des infizierten Organismus.[2]
Genom
Das Genom fast aller Influenzaviren besteht aus acht RNA-Abschnitten (Segmenten) negativer Polarität. Diese acht RNA-Moleküle enthalten die genetische Information, die für die Vermehrung und den Zusammenbau der Viruspartikel benötigt wird. Die Segmentierung des Genoms ist auch für die erhebliche Steigerung der genetischen Veränderlichkeit (Variabilität) der Influenzaviren über eine hohe Mutationsfrequenz und die Fähigkeit zur genetischen Reassortierung verantwortlich.
Die Segmente kodieren bis zu elf virale Proteine[3] : Hämagglutinin (HA), Neuraminidase (NA), Nukleoprotein (NP), die Matrixproteine (M1) und (M2), die Polymerase Proteine (PB1), (PB2) und (PA) und die Nichtstrukturproteine (NS1) und (NS2). Eines der acht Gensegmente enthält allein das NS-Gen, welches die beiden Nichtstrukturproteine (NS1) und (NS2) bildet. Ein Forscherteam um Clayton Naeve vom St. Jude Children's Hospital in Memphis, Tennessee - USA hat durch vergleichende Analysen der DNA-Sequenzen von Influenzaviren festgestellt, dass sich an einem Ende des NS-Gens ein Abschnitt befindet, der vermutlich mit über die Heftigkeit eines Infektionsverlaufs entscheidet. Änderungen in diesem Genabschnitt führen zu einer Variation in einem variablen Bereich auf dem NS1-Protein. Dieser variable Proteinbereich kann sich je nach Struktur mehr oder minder effektiv an sogenannte PDZ-Domänen (spezieller Teilbereich von Eiweißmolekülen in Zellen) binden und dadurch die Signalübermittlung in den Zellen unterschiedlich stark stören. Eine Störung der Signalübermittlung bewirkt dann eine Überstimulation des Immunsystems, bei der viele Entzündungsbotenstoffe ausgeschüttet werden.
Der hiermit angesprochene Genomabschnitt ist demzufolge auch als einer der Orte anzusehen, bei denen eine Anpassung der verschiedenen Gattungen und Virussubtypen an ihre jeweiligen Reservoirwirte stattfindet oder schon stattgefunden hat.
Vermehrung
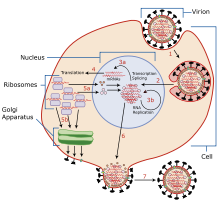
Die Influenzaviren vermehren (replizieren) sich beim Menschen im Atemtrakt (Respirationstrakt) eines infizierten Individuums. Menschliche Grippeviren bevorzugen gemäß IMPP Zellen des Flimmerepithels. Im Gegensatz dazu vermehrt sich das Grippe-Virus bei Vögeln hauptsächlich in den Darmepithelzellen.[4]
Die Viren wandern bei ihrem Wirt durch das Mucin in die Epithelzellen, die als Wirtszellen dienen. Dafür dass sie dabei nicht mit dem Schleim verkleben, sorgt die Neuraminidase.[5] In einer einzigen Wirtszelle können sich bis zu 100.000 neue Influenzaviren bilden, bevor diese dann abstirbt und anschließend die freigesetzten Viren weitere Nachbarzellen infizieren. Diese produzieren dann ebenfalls im Schneeballsystem jeweils hunderttausende neue Viren. So erklärt sich auch die Schnelligkeit, mit der sich in der Regel diese virale Infektion im Körper eines Betroffenen ausbreitet.
Systematik
Es gibt drei verschiedene Gattungen dieser Viren, welche mit den Gattungen Thogotovirus und Isavirus alle zusammen zur Familie der Orthomyxoviren gehören.
- Influenza-A-Viren
- Die lineare, einzelsträngige RNA ihres Genoms besitzt acht Segmente und sie zeichnen sich besonders durch große Unterschiede in den antigenen Eigenschaften aus, die im Vergleich zu den anderen Gattungen auf besonders hoher Mutationsfrequenz und Neugruppierungen beruhen. Diese Untertypen befallen üblicherweise jeweils nur bestimmte Wirte. Dazu zählen der Mensch und verschiedene Säugetierarten wie Schwein (siehe unter Schweineinfluenza), Pferd (siehe unter Pferdeinfluenza), Nerz, Seehund und Wal, zahlreiche Vogelarten und seit kurzem auch der Haushund. Das primäre Reservoir aller Influenza-A-Viren liegt im Wassergeflügel.
- Influenza-B-Viren
- Ihr Genom hat ebenfalls eine acht-fach segmentierte lineare, einzelsträngige RNA und sie befallen nur Menschen.
- Influenza-C-Viren
- Im Gegensatz zu den Influenza-A- und B-Viren hat die lineare, einzelsträngige RNA des Genoms der Influenza-C-Viren nur sieben Segmente und sie besitzen keine Neuraminidase (NA). Außerdem liegt bei diesen Viren ein Glykoprotein [Oberflächen-Haemagglutinin-Esterase-Fusion-Protein (HEF)] vor, das sowohl die Aufgaben der Rezeptorbindung des Virus an die Wirtszelle, der anschließenden Eindringung (Fusion) wie auch der späteren Freisetzung der neugebildeten Viren aus der Zelle übernimmt. Dieser Virus-Typ C befällt Mensch und Schwein (siehe ebenfalls unter Schweineinfluenza), doch spielt er bei Erkrankungen des Menschen keine relevante Rolle, da er wenn überhaupt nur zu milden Erkrankungen führt.
In Fachkreisen wird jeder Virusstamm mit den Kennungen Typus, Ort der erstmaligen Isolierung (Virusanzucht), Isolierungsnummer, Isolierungsjahr (Beispiel: Influenza B/Shanghai/361/2002) und nur bei den A-Viren auch zusätzlich mit der Kennung des Oberflächenantigens benannt [Beispiel: Influenza A/California/7/2004 (H3N2)].
Variabilität und Subtypen
Antigendrift
Eine Häufung von Punktmutationen in den Nukleotiden führt zu einer Veränderung der Erbinformationen (Gendrift). Betreffen solche Veränderungen die beiden Glykoproteine HA und NA (bzw. den sie kodierenden Bereich) bewirkt dies eine Änderung der Oberflächenantigene des Grippevirus (Antigendrift). Menschliche Antikörper können immer nur eine solche Variante erkennen. Diese eher kleinen Veränderungen sind der Grund dafür, dass ein Mensch mehrmals in seinem Leben mit einer anderen nur geringfügig veränderten Virusvariante (Driftvariante) infiziert werden kann und dass sowohl Epidemien wie regional begrenzte Ausbrüche regelmäßig wiederkehren.
- → Siehe auch: Antigenerbsünde
Antigenshift bei Doppelinfektion
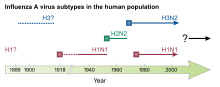
Wird ein Organismus gleichzeitig von zwei Virusvarianten infiziert (Doppelinfektion), kann es zu einer Neuzusammenstellung (zum Neuarrangement) unter den zweimal acht Genomsegmenten der beteiligen Influenzaviren kommen, in dem einzelne oder mehrere RNA-Moleküle zwischen den Influenzaviren in einer doppelt infizierten Zelle ausgetauscht werden. Diesen Vorgang nennt man genetische Reassortierung, und er kann im Menschen, aber auch in anderen Wirten, wie beispielsweise bei Vögeln und Schweinen erfolgen. Die so verursachten größeren als Antigenshift bezeichnete Veränderungen in den viralen Oberflächenantigenen werden allein bei den Influenza-A-Viren beobachtet (Shiftvarianten), allerdings kommen sie nur selten vor. Derartige Veränderungen können dann der Ursprung von Pandemien sein, von denen es im 20. Jahrhundert die von 1918 bis 1919 mit dem Subtyp H1N1, 1957 mit H2N2, 1968 mit H3N2 und die von 1977 mit dem Wiederauftauchen von H1N1 gab.
Influenza-A-Subtypen
Im Allgemeinen werden die Influenza-A-Viren in erster Linie nach bestimmten, deutlich unterschiedlichen Oberflächeneigenschaften in Untertypen bzw. Subtypen eingeteilt. Dies geschieht nach dem Muster A/HxNx oder A/Land/HxNx/Probe. Bisher wurden bei ihnen insgesamt 16 H-Untertypen und 9 N-Untertypen erkannt.
Die wichtigsten Oberflächenantigene beim Influenza-A-Virus sind die Hämagglutinine (H1, H2, H3, H5, seltener H7 und H9) und die Neuraminidase (N1, N2, seltener N7), und deshalb sind auch folgende Subtypen für den Menschen von besonderer Bedeutung:
A/H1N1
A/H1N1 ist ein häufig umlaufender Subtyp der Humaninfluenza. Er kann besonders leicht in menschliche Körperzellen eindringen und sein Erbgut einschleusen. Da sein erster Nachweis 1930 aus Schweinen erfolgt war,[6][7] werden durch diesen Subtyp verursachte Infektionen beim Schwein als Schweineinfluenza und beim Menschen – bezogen auf eine bis 2009 unbekannte Variante – umgangssprachlich als „Schweinegrippe“ bezeichnet.
Eine Variante von A/H1N1 konnte als Auslöser der so genannten Spanischen Grippe von 1918/1920 im Lungengewebe von Opfern nachgewiesen werden. 2005 gelang Jeffery Taubenberger eine Rekonstruktion des Erregers der Spanischen Grippe aus Genfragmenten. Im Jahre 2007 wurde durch Forscher des St. Jude Children's Hospital, Memphis (Tennessee), bekannt, dass ein nur 90 Aminosäuren großes Virusprotein mit der Bezeichnung PB1-F2 verantwortlich zu sein scheint für die ungewöhnlich hohe Letalität von A/H1N1 in den Jahren nach 1918. [8] Es bewirke besonders ausgeprägte Entzündungen bei den Infizierten. Testtiere erkrankten schon dann schwer, wenn nur ihre Nasenschleimhaut mit dem Protein in Berührung kam. Die heute noch kursierenden H1N1-Viren verfügen hingegen über ein verstümmeltes, nur 67 Aminosäuren umfassendes Protein: Dies sei die Folge davon, dass infolge einer Mutation ein Stopp-Signal ins PB1-F2-Gen eingefügt worden sei, so dass es nicht mehr vollständig abgelesen werde und das entstehende Protein daher minder pathogen sei.
Ein erneuter weltweiter Ausbruch – die so genannte Russische Grippe – ereignete sich 1977.[9]
Ende Januar 2008 wurde von norwegischen Ärzten bei normalen Grippepatienten ein gegen Oseltamivir resistenter Virusstamm (A/H1N1-H274Y) entdeckt,[10] der sich inzwischen weltweit verbreitet hat.[11] Mitte 2009 sind weltweit 96%, in den entwickelten Ländern mit Benutzung von Oseltamivir praktisch alle[12] saisonalen A/H1N1-Viren resistent.[13]
Im April 2009 ereignete sich in Mexiko ein epidemieartiger Ausbruch einer bis dahin unbekannten Variante des H1N1-Subtyps, an dem zahlreiche Menschen erkrankten (siehe: Pandemie H1N1 2009).
A/H2N2
Ein weltweiter Ausbruch dieses Subtyps der Humaninfluenza war 1957 die Ursache einer Pandemie, die als Asiatische Grippe bezeichnet wurde.
A/H3N2
A/H3N2 ist in Europa und in den USA verbreitet. Ein weltweiter Ausbruch dieses Subtyps der Humaninfluenza (Fujian Typ) war 1968 die Ursache einer Pandemie, die als Hongkong-Grippe bezeichnet wurde.
A/H3N8
A/H3N8 war ursprünglich bei Pferden verbreitet, ging jedoch im frühen 21. Jahrhundert auf Hunde über. Es kommt bei dieser Art vor allem in den USA vor.[14] Infektionen beim Menschen sind bislang keine bekannt.
A/H5N1
Der Subtyp A/H5N1 ist einer von mehreren Auslösern der Geflügelpest. Trotz mehrerer Dutzend Übergänge auch auf den Menschen gehört dieser (HPAI, Highly Pathogenic Avian Influenza) Subtyp bisher nicht in die Reihe der Influenza-A-Viren, die beim Menschen eine Grippe auslösen können, da das Virus bislang allenfalls in sehr seltenen Einzelfällen von Mensch zu Mensch übertragbar ist. Die WHO hält es zur Zeit noch für wenig wahrscheinlich, dass sich das Virus an den Menschen anpassen und eine Pandemie auslösen könnte.
A/H5N2
Dieser Subtyp trat u. a. im Sommer 2005 in Japan auf, weswegen Presseberichten zufolge mehr als 1,5 Millionen Hühner und anderes Geflügel getötet wurden.
A/H5N3
Dieser Subtyp verursachte im Jahr 1961 in Südafrika ein großes Sterben unter frei lebenden Seeschwalben, was der erste Nachweis von Influenza-Viren in einer Wildvogel-Population war.
A/H7N2
2002 wurde dieses Virus in den USA erstmalig auf den Menschen übertragen.
A/H7N3
A/H7N3 wurde zuerst bei Truthähnen in Großbritannien im Jahre 1963 entdeckt.
In Kanada wurde die Präsenz von H7N3 in mehreren Putenfarmen in British Columbia im Februar 2004 nachgewiesen, 18 Farmen wurden unter Quarantäne gestellt. Zwei Menschen infizierten sich hier, beide überstanden die Infektion ohne Folgen. Die Symptome sind ähnlich der einer leichten Grippe.[15][16]
In Großbritannien wurde H7N3 im April 2006 gefunden. Es infizierte Puten und einen Mitarbeiter eines Zuchtbetriebs in der Stadt North Tuddenham.
A/H7N7
Dieser Subtyp gehört nicht in die Reihe der Influenza-A-Viren, die beim Menschen eine Grippe auslösen können. Zuletzt 2003 wurden in den Niederlanden auch 89 Infektionen von Menschen mit diesem (HPAI, Highly Pathogenic Avian Influenza) Subtyp bestätigt. Ein Fall verlief tödlich. Das Virus kann auch Pferde befallen.
→ Siehe auch: Geflügelpest
A/H9N2
Dieser Subtyp gehört nicht in die Reihe der Influenza-A-Viren, die beim Menschen eine Grippe auslösen können, und er wurde bislang auch beim Menschen nur in einer minder pathogenen Form (LPAI, Lowly Pathogenic Avian Influenza) von Peiris et al. (1999) isoliert und dokumentiert. Bei drei Fällen in Hongkong und China (1999, 2003) erholten sich die Patienten von dieser influenzaähnlichen Infektion.
Influenza-B-Subtypen
Die Influenza-B-Viren werden in zwei Stamm-Linien eingeteilt
- B/Victoria-Linie
- B/Yamagata-Linie
Influenza-C-Subtypen
Die Unterschiede zwischen einzelnen Virusstämmen sind derart gering, dass hier bislang keine weitere Unterteilung vorgenommen wurde.
Umweltstabilität
Je nach Temperatur ist die Umweltstabilität der Influenzaviren sehr unterschiedlich. Bei einer normalen sommerlichen Tagestemperatur von etwa 20 °C können an Oberflächen angetrocknete Viren in der Regel zwei bis acht Stunden überdauern. Bei 22 °C überstehen sie sowohl in Exkrementen wie auch in Geweben verstorbener Tiere und in Wasser mindestens vier Tage, bei einer Temperatur von 0 °C mehr als 30 Tage und im Eis sind sie nahezu unbegrenzt überdauerungsfähig. Forscher der NIH in den USA stellten 2008 fest, dass die Lipidhülle des Influenza-Virus sich bei tieferen Temperaturen verändert. Damit wird das Virus stabilisiert und bleibt länger pathogen.[17]
Oberhalb von 22 °C verringert sich allerdings die Umweltstabilität der Influenzaviren sehr deutlich. Bei 56 °C werden sie innerhalb von 3 Stunden und bei 60 °C innerhalb von 30 Minuten inaktiviert.[18] Ab 70 °C verliert das Virus endgültig seine Infektiosität.
Vorkommen
Die Influenzaviren und die durch sie ausgelösten Erkrankungen existieren weltweit, allerdings kommen im Gegensatz zu den anderen Virustypen die Influenza-C-Viren nur gelegentlich vor.
Einzelnachweise
- ↑ a b Zhi-Yong Yang, Chih-Jen Wei, Wing-Pui Kong, Lan Wu, Ling Xu, David F. Smith, Gary J. Nabel: Immunization by Avian H5 Influenza Hemagglutinin Mutants with Altered Receptor Binding Specificity. In: Science. 317, Nr. 5837, 10. August 2007, ISSN 0036-8075, S. 825 - 828, doi:10.1126/science.1135165.
- ↑ http://www.berlinonline.de/berliner-zeitung/archiv/.bin/dump.fcgi/2006/1104/wissenschaft/0097/index.html
- ↑ http://www.nature.com/nature/journal/v463/n7282/full/nature08699.html Renate König et al.: Human host factors required for influenza virus replication, Nature 463, 11 Februar 2010, S. 813-817, doi:10.1038/nature08699
- ↑ Univ.-Prof. Mag.pharm. Dr. Franz X. Heinz, Dem Geheimnis der Influenza Pandemien auf der Spur, abgerufen 18. November 2009
- ↑ Der Lebenszyklus des Influenza-Virus - eine erste Übersicht, abgerufen, 18. November 2009
- ↑ Richard E. Shope: Swine Influenza: III. Filtration and Ion Experiments and Etiology. The Journal of Experimental Medicine, Band 54, 1931, S. 373–385
- ↑ Suburban Emergency Management Project, Biot Report 162 vom 9. Januar 2005: „What Is Swine Flu?“
- ↑ Julie L. McAuley u.a.: Expression of the 1918 Influenza A Virus PB1-F2 Enhances the Pathogenesis of Viral and Secondary Bacterial Pneumonia. Cell Host and Microbe 2, S. 240-249, 11. Oktober 2007, doi:10.1016/j.chom.2007.09.001, [1]
- ↑ http://www.labor-limbach.de/Informationen_zur_Vo.317.0.html
- ↑ http://www.ecdc.europa.eu/en/Health_Topics/influenza/news/news_Influenza_080131.aspx##Publichealth_1 PH DEVELOPMENTS – SEASONAL INFLUENZA – ANTIVIRALS; Resistance to Oseltamivir (Tamiflu) found in some European influenza virus samples
- ↑ http://www.who.int/csr/disease/influenza/H1N1webupdate18072008.pdf WHO: Influenza A(H1N1) virus resistance to oseltamivir - 2008 influenza season, southern hemisphere. 18 July 2008
- ↑ http://www.who.int/entity/csr/disease/influenza/Global_H1N1Resistance_20090528.png
- ↑ http://www.who.int/csr/disease/influenza/2008-9nhemisummaryreport/en/ WHO: Influenza A virus resistance to oseltamivir and other antiviral medicines
- ↑ T Jamanaka et al. (2009): "Interspecies transmission of equine influenza virus (H3N8) to dogs by close contact with experimentally infected horses" Vet Microbiol, epub ahead of print, PMID 19596528
- ↑ S. Aleina Tweed, Danuta M. Skowronski, Samara T. David, Andrew Larder, Martin Petric, Wayne Lees et. al.: Human Illness from Avian Influenza H7N3, British Columbia. In: Emerging Infectious Diseases. 10, Nr. 12, November 2004, ISSN 1080-6059 (online hier, abgerufen am 27. Juni 2009).
- ↑ Martin Hirst, Caroline R. Astell, Comments Malachi Griffith, Shaun M. Coughlin, Michelle Moksa, Thomas Zeng et. al.: Novel Avian Influenza H7N3 Strain Outbreak, British Columbia. In: Emerging Infectious Diseases. 10, Nr. 12, November 2004, ISSN 1080-6059 (online hier, abgerufen am 27. Juni 2009).
- ↑ Ivan V. Polozov, Ludmila Bezrukov, Klaus Gawrisch und Joshua Zimmerberg: Progressive ordering with decreasing temperature of the phospholipids of influenza virus. In: Nature Chemical Biology, Band 4, 2008, S. 248–255, PMID 18311130
- ↑ Akademie für öffentliches Gesundheitswesen Düsseldorf (Hrsg.): Blickpunkt öffentliche Gesundheit 04/2001
Weblinks
Kategorien:- Viren, Viroide und Prionen
- Meldepflichtiger Erreger
Wikimedia Foundation.