- Sekundärstruktur
-
Die Sekundärstruktur von Makromolekülen beschreibt die konformationelle Anordnung der Backbonesegmente – wie einer Polypeptidkette eines Proteins – ohne die Konformation der Seitenkette oder ihr Verhältnis zu anderen Segmente mit zu betrachten. Bei so komplexen Molekülstrukturen wie Proteinen definiert die Sekundärstruktur insbesondere charakteristische lokale Strukturelemente wie Helices oder Faltblätter.[1]
Sie ist bestimmt durch die von Wasserstoffbrücken zwischen einzelnen Elementen definierte Topologie,[2] sowie durch die Primärstruktur. Manchmal – etwa bei Polysacchariden – spielt auch die Form der Polymerisierung eine Rolle.
Die Darstellung der Sekundärstruktur, oder des Polymerrückgrats mit den Sekundärstrukturelementen, bietet einen besseren Überblick als die Darstellung der vollständigen Molekülstruktur. Andererseits gibt sie einen wesentlich genaueren Einblick in die tatsächliche Struktur als die Abbildung durch Pauli-Schreibweise oder Fischer-Projektion.
In der RNA- und Proteinstrukturvorhersage bildet die Vorhersage der Sekundärstrukturelemente ein wichtiges, und im Vergleich weniger schwieriges Unterproblem.
Die genaue Anordnung der Atome im dreidimensionalen Raum, welche sämtliche physikalischen Wechselwirkungen innerhalb des Moleküls und mit seiner Umgebung einbezieht, wird dagegen als Tertiärstruktur bezeichnet. Die Einteilung zu einer Hierarchie in Primärstruktur, Sekundärstruktur, Tertiärstruktur und Quartärstruktur wurde 1952 durch Kaj Ulrik Linderstrøm-Lang vorgeschlagen.
Inhaltsverzeichnis
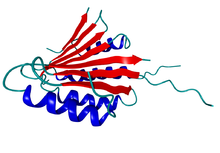
Sekundärstruktur von Proteinen
Bei Proteinen ist die Sekundärstruktur bestimmt durch Wasserstoffbrücken zwischen den CO- und NH-Gruppen des Peptidrückgrats. Dafür gibt es zwar unüberschaubar viele Möglichkeiten, jedoch zeigt sich, dass einige Motive, oder Sekundärstrukturelemente, besonders häufig vorkommen:
Bereiche, die keine definierte Sekundärstruktur aufweisen, werden als Random-coil bezeichnet.
Mit Ausnahme der Beta-Schleifen und Random-coils zeichnen sich diese Bereiche dadurch aus, dass in ihnen die beiden einzig möglichen Drehwinkel ψ und φ des Peptidrückgrates festgelegt sind und sich über die Länge des Sekundärstrukturelementes hindurch periodisch wiederholen. Im Ramachandran-Plot sind die möglichen Sekundärstrukturen als Funktion der zugehörigen ψ/φ Winkelpaare dargestellt. Durch die H-Brücken innerhalb des Peptidrückgrates werden die Sekundärstrukturelemente energetisch stabilisiert. Je nach Art der Sekundärstruktur können bestimmte Aminosäureseitenketten auf deren Struktur destabilisierend wirken.
In ein und demselben Protein liegt meistens eine Mischung aus den verschiedenen Sekundärstrukturelementen vor. Die vollständige Proteinstruktur (d.h. die Abfolge bzw. Anordnung der Sekundärstrukturelemente) wird als Tertiärstruktur bezeichnet. Sie ist für jedes Protein charakteristisch und für die biologische Funktion unbedingt notwendig. Während bzw. nach der Herstellung des Proteins durch Translation eines RNA-Moleküls wird das Protein durch Proteinfaltung in die biologisch wirksame Form überführt. Dieser Vorgang wird u.a. durch Chaperone unterstützt.
Sekundärstruktur von Nukleinsäuren
Auch Nukleinsäuren, also DNA und RNA, können Sekundärstrukturen bilden. Voraussetzung hierfür ist, dass das Nukleinsäuremolekül zunächst als Einzelstrang vorliegt. Komplementäre Abschnitte können dann Wasserstoffbrücken und intramolekulare Doppelstränge bilden, während nicht komplementäre Abschnitte einzelsträngig bleiben. Sekundärstrukturen von Nukleinsäuren bezeichnet man in Anlehnung an ihre Form als Haarnadelstruktur (hairpin loop) wenn ein gepaarter doppelsträngiger Abschnitt (Stamm) einen kurzen einzelsträngigen Abschnitt (kleine Schleife) oder als stem-loop, wenn ein gepaarter doppelsträngiger Abschnitt (Stamm) einen längeren einzelsträngigen Abschnitt (große Schleife) einschließt.
Die Sekundärstrukturen haben wichtige Funktionen bei der Regulation der Transkription, sie können als Primer dienen (siehe Telomerase) oder Voraussetzung für die enzymatische Aktivität der Ribosomen sein (siehe rRNA).
Algorithmen zur RNA-Strukturvorhersage
Siehe auch
Einzelnachweise
- ↑ Eintrag: secondary structure. In: IUPAC Compendium of Chemical Terminology (the “Gold Book”). doi:10.1351/goldbook.S05530.
- ↑ Hermann J. Roth, Christa E. Müller, Gerd Folkers: Stereochemie und Arzneistoffe, Wissenschaftliche Verlagsgesellschaft Stuttgart, 1998, S. 240−244, ISBN 3-8047-1485-4.
Wikimedia Foundation.