- Papillomaviridae
-
Papillomaviridae Systematik Reich: Viren Ordnung: nicht klassifiziert Familie: Papillomaviridae Taxonomische Merkmale Genom: dsDNA zirkulär Baltimore: Gruppe 1 Symmetrie: ikosaedrisch Hülle: keine Wissenschaftlicher Name Papillomaviridae (engl.) Links NCBI Taxonomie: 151340 ICTVdB Virus Code: 00.099 Die Virusfamilie Papillomaviridae umfasst 16 Gattungen von unbehüllten Viren mit einer doppelsträngigen, zirkulären DNA als Genom. Bis 2002 bildete die Familie Papillomaviridae als Gattung Papillomavirus zusammen mit der jetzt eigenen Familie Polyomaviridae die Familie Papovaviridae. 1964 hatte Joseph Melnick aufgrund der morphologischen und genetischen Ähnlichkeiten diese heute obsolete Virusfamilie vorgeschlagen. Die Vermehrungsstrategie, das Spektrum der Erkrankungen und die Anordnung der Gene sind jedoch so unterschiedlich, dass man sie in zwei verschiedene Familien unterteilte.
Papillomviren verursachen bei sehr vielen verschiedenen Wirbeltieren Hautwucherungen im Sinne von Warzen (Papillome). Das erste als Virus erkannte Agens aus dieser Gruppe wurde von Richard Shope 1932 bei erkrankten Kaninchen isoliert (Shopes Kaninchenpapillomvirus). Die Entstehung bösartiger Tumoren aus diesen gutartigen Wucherungen wurde bereits damals beobachtet. Heute ist die Entstehung verschiedener Krebsarten bei Menschen und Tieren mit Vertretern der Papillomaviridae verbunden. Beim Menschen ist dies vorwiegend das Cervixkarzinom durch Infektionen mit Humanen Papillomviren. Die Vermehrung von Papillomviren ist strikt auf Epithelien beschränkt und sie bedürfen zur Vermehrung Zellen, die sich in einer Phase der Entdifferenzierung befinden. Aufgrund dieses strengen Gewebstropismus ist bislang eine vollständige Vermehrung der Papillomaviridae in Zellkulturen nicht möglich.
Inhaltsverzeichnis
Morphologie
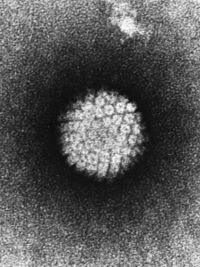
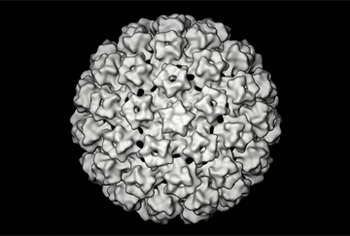
Die etwa 55 nm großen, unbehüllten Virionen der Papillomviren bestehen aus einem ikosaedrischen Kapsid (T=7), das aus 72 Kapsomeren aufgebaut ist.[1] Von den Kapsomeren sind 60 in einer fünfstrahligen Symmetrie (Pentamere) und diese wiederum in 12 sechsstrahligen (Hexamere) angeordnet.[2] Zwischen den Pentameren bestehen Disulfidbrücken. Bei fehlerhafter Reifung und Zusammenbau können auch filamentöse Kapside beobachtet werden. Die Kapsomere werden von zwei Strukturproteinen (L1 und L2) gebildet. Das L1 (55 bis 60 kDa) ist mit über 80 % Gewichtsanteil dominierend. Es besitzt nach außen gerichtete, speziesspezifische Epitope. Das L2 (50 bis 53 kDa) besitzt gruppenspezifische Epitope. Die Virionen besitzen eine hohe Umweltstabilität; milde Detergenzien (Seife), saure pH-Werte und zur Virusinaktivierung üblicherweise verwendete fettlösende Substanzen (beispielsweise 2-Propanol) inaktivieren Papillomviren nicht. Sie sind für 1 Stunde bei 50 °C hitzestabil.
Im Inneren der Kapside befindet sich die zirkuläre DNA des Virusgenoms. Diese ist mehrfach verdrillt („supercoiled“) und bildet zusammen mit zellulären Histonen einen Nukleoproteinkomplex, der den eukaryotischen Nukleosomen strukturell sehr ähnelt. Von den fünf bekannten Histonen findet man die Histone H2a, H2b, H3 und H4.
Genomorganisation
Das Genom der Papillomviren ist etwa 6800 bis 8400 bp groß und kodiert mit 9 bis 10 Offene Leserahmen (ORF) für die frühen Virusproteine (early: E1-E8) und die späten Strukturproteine (late: L1 und L2). Bei einigen Spezies fehlen E3- oder E8-Leserahmen. Im Gegensatz zu den Polyomaviridae ist die Leserichtung für alle ORFs gleich. Vor den Genen für die frühen Virusproteine befindet sich eine regulatorische Region (LCR, long control region), die Steuerungssequenzen (Promotoren, Enhancer und Replikationsursprung) enthalten. Die Leserahmen überlappen sich teilweise und liegen in verschiedenen Leserastern. Die im Zellkern durch zelluläre RNA-Polymerasen transkribierten viralen mRNAs unterliegen weiteren Modifizierungen wie einem Capping, einer Polyadenylierung und einem RNA-Splicing. Zur Vermehrung der genomischen DNA der Papillomaviridae besitzen die Viren keine eigene DNA-Polymerase. Sie sind bei der Virusreplikation darauf angewiesen, dass ausreichende Mengen zellulärer DNA-Polymerasen vorhanden und aktiv sind. Da dies besonders während der Zellteilung der Fall ist, haben die Papillomaviridae verschiedene Mechanismen entwickelt, um die Wirtszelle zur ständigen Teilung anzuregen. Diese Mechanismen sind die Ursache für die typischen Gewebswucherungen bei Infektionen mit Papillomviren und eine mögliche Entartung der Zellen.
Systematik
-
- Familie Papillomaviridae
-
- Genus Alphapapillomavirus
- Genus Betapapillomavirus
- Genus Gammapapillomavirus
- Genus Deltapapillomavirus
- Genus Epsilonpapillomavirus
- Genus Zetapapillomavirus
- Genus Etapapillomavirus
- Genus Thetapapillomavirus
- Genus Iotapapillomavirus
- Genus Kappapapillomavirus
- Genus Lambdapapillomavirus
- Genus Mupapillomavirus
- Genus Nupapillomavirus
- Genus Xipapillomavirus
- Genus Omikronpapillomavirus
- Genus Pipapillomavirus
nicht klassifizierte Spezies innerhalb der Familie Papillomaviridae:
-
-
-
- Spezies Trichosurus-vulpecula-Papillomvirus
- Spezies Opossum-Papillomvirus
-
-
Quellen
- E.-M. de Villiers et al.: Family Papillomaviridae. In: C. M. Fauquet, M. A. Mayo et al.: Eighth Report of the International Committee on Taxonomy of Viruses. London, San Diego, 2005 S. 239ff ISBN 0-12-249951-4
- Peter M. Howley, Douglas R. Lowy: Papillomaviruses. In: David M. Knipe, Peter M. Howley (eds.-in-chief): Fields’ Virology. 5. Auflage, Band 2, Philadelphia 2007, S. 2299ff ISBN 0-7817-6060-7
Einzelnachweise
- ↑ T. S. Baker et al.: Structures of bovine and human papillomaviruses. Analysis by cryoelectron microscopy and three-dimensional image reconstruction. Biophys. J. (1991) 60(6): S. 1445-1456 PMID 1663794 (Volltext)
- ↑ Y. Modis et al.: Atomic model of the papillomavirus capsid. EMBO Journal (2002) 21(18): S. 4754-4762 PMID 12234916 (Volltext)
Weblinks
- Molekularer Aufbau des Papillomvirus-Kapsids (EMBO 2002)
- Phylogenetische Verwandtschaft der Papillomviren und Abgrenzung der Gattungen (J. Gen. Virol 2007)
-
Wikimedia Foundation.