- T-RNA
-
tRNA ist die Kurzform für transfer-RNA. Transfer-RNAs sind kurze Ribonukleinsäuren (RNA). Die Länge reifer tRNAs liegt in der Regel zwischen 73 und 95 Nukleotiden. Sie vermitteln bei der Translation die richtige Aminosäure zum entsprechenden Codon auf der mRNA.
Inhaltsverzeichnis
Struktur
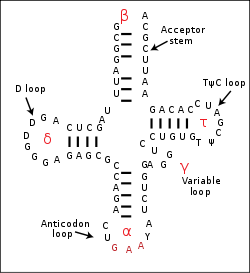
In jedem tRNA-Molekül treten Paarungen konjugierender Basen (Adenin und Uracil; Cytosin und Guanin) auf. Sie sind die Ursache für die kleeblattartige Sekundärstruktur der tRNA. In den Schleifen der tRNA-Arme können sich die vorliegenden Basen jedoch nicht durch Wasserstoffbrückenbildung zusammenschließen, da sie nicht konjugierend sind.
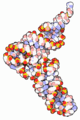
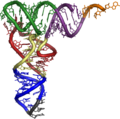
Die tatsächliche dreidimensionale Tertiärstruktur ähnelt jedoch einem L, dessen zwei Arme den Aminosäuren-Akzeptorstamm und die Anticodonschleife bilden. Die funktionell wichtigen Abschnitte liegen also so weit wie möglich voneinander entfernt.
Auf der Anticodonschleife befindet sich ein spezifisches Basentriplett, das so genannte Anticodon.
In der Kleeblattstruktur befindet sich links die so genannte Dihydrouracil-Schleife. Diese dient vor allem der Erkennung der tRNA durch die Aminoacyl-tRNA-Synthetase. Auf der rechten Seite der Kleeblattstruktur ist die T(Psi)C-Schleife. Dabei steht das Psi für Pseudouridin. Diese Schleife/dieser Arm bindet an die 5S-rRNA der großen Ribosomenuntereinheit.
Die dreidimensionale Struktur von tRNA wurde bereits 1974 unabhängig von zwei Gruppen um Rich und Klug aufgeklärt.[1][2]
Funktion
Bei der Translation in der Proteinbiosynthese muss im Ribosom entsprechend des genetischen Codes zu jedem Basentriplett auf der mRNA die passende Aminosäure an die Peptidkette angehängt werden. Diese Aufgabe wird durch die tRNA vermittelt. Dazu gibt es für jede Aminosäure mindestens eine, häufig aber auch mehrere verschiedene tRNAs. In der Regeln werden diese abhängig von ihrer Sequenz unter ATP-Verbrauch von der jeweiligen Aminoacyl-tRNA-Synthetase am 3’-Ende spezifisch mit der zugehörigen Aminosäure beladen. Dazu wird an die 3’-Hydroxygruppe der Ribose des Adenosins die Carboxygruppe der Aminosäure in Esterbindung angehängt und eine Aminoacylgruppe entsteht. Häufig erkennt die Aminoacyl-tRNA-Synthetase dazu das Anticodon auf der tRNA. Es können aber auch anderer Sturkurelemente bei der Erkennung ein Rolle spielen, hauptsächlich der Akzeptorstamm.
Zunächst wurde angenommen, dass in allen Organismen für jede Aminosäure genau eine Aminoacyl-tRNA-Synthetase existiert, die alle zur entsprechenden Aminosäure gehörenden tRNAs beladen kann. Später wurde aber entdeckt, dass einigen Organismen eine oder mehrere Aminoacyl-tRNA-Synthetasen fehlen, diese aber dennoch die entsprechenden Aminosäuren in ihre Proteine einbauen können und auch die nötigen tRNAs besitzen. Diese müssen also einen anderen Mechanismus zur Beladung dieser tRNAs besitzen. So fehlt in vielen Archaea, Bakterien, Chloroplasten und Mitochondrien eine Aminoacyl-tRNA-Synthetase für Glutamin. Stattdessen bindet die Aminoacyl-tRNA Synthetase für Glutamat dieses sowohl an tRNA für Glutamat als auch an tRNA für Glutamin. Das „fehlerhaft“ an die tRNA für Glutamin gebundene Glutamat wird anschließend durch eine Transamidase in Glutamin umgewandelt.
Die aminoacylierten tRNAs werden von den Ribosomen für die Proteinbiosynthese genutzt. Passt das Anitcodon zum entsprechenden Basencodon der messenger-RNA (mRNA), so kann sich die tRNA dort anlagern, und die herantransportierte Aminosäure an das entstehende Protein anknüpfen.
Entsprechend des genetischen Codes müsste für jedes Basentriplett, das für eine Aminosäure und nicht für ein Stopcodon kodiert ist, eine tRNA existieren – in der Regel also 64.[3] Es wurde aber festgestellt, dass die Zahl der tRNA davon deutlich nach unten abweicht. Die genaue Anzahl unterscheidet sich in den Organismen, jedoch sind es nicht mehr als 41. Dennoch werden in allen Organismen alle Basentripletts zur Proteincodierung verwendet. Diese Abweichung wird durch die Wobble Theorie erklärt. Demnach sind durch die Tatsache, dass der genetische Code ein degenerierter Code ist, bei dem mehrere Basentripletts für die gleiche Aminosäure kodieren können, häufig nur zwei Basen für die Erkennung nötig, da sich Basentripletts für die gleiche Aminosäure oft nur in einer Base unterscheiden.
tRNA-Gene
Organismen variieren in der Anzahl ihrere tRNA-Gene auf ihrem Genom. Der Fadenwurm Caenorhabditis elegans, ein typischer Modellorganismus für genetische Studien, besitzt 29.647[4] Gene in seinem Zellkern-Genom, wobei 620 davon für tRNA kodieren.[5][6] Die Backhefe besitzt 275 tRNA-Gene in ihrem Genom.
In Prokaryonten sind meist mehrere Gene in Operons zusammengefasst. Die Gene können für Proteinsequenzen oder verschiedene RNA-Produkte kodieren, darunter auch tRNA. In der Regel werden so mehrere tRNA auf einem Operon zusammengefasst und diese können auch proteinkodierende Gene enthalten.
Bei Eukaryonten muss zwischen tRNA-Genen auf der DNA im Zellkern und tRNA-Genen auf der DNA von Mitochondrien oder Plastiden wie Chloroplasten unterschieden werden. In Plastiden sind die Gene ähnlich wie bei Prokaryonten in Operons organisiert. Die Ausstattung mit tRNA-Genen (z. B. 30 tRNA-Gene bei Marchantia polymorpha) beinhaltet alle tRNA-Gruppen, die für die Proteinsynthese in Plastiden erforderlich sind.
Transkription der tRNA
Prokaryonten besitzen nur eine RNA-Polymerase. Diese transkribiert den Leserahmen einen Operons, so dass in der Regel eine polycistronische mRNA entsteht. Aus dieser müssen ggf. die tRNAs durch endonukleolytische Schnitte in den intercistronischen Bereichen herausgelöst werden.
In Eukaryonten wird die tRNA im Zellkern durch RNA-Polymerase III transkribiert[7]. Proteinkodierende Gene werden dort hingegen durch RNA-Polymerase II transkribiert. Die tRNA in Plastiden wird durch eine eine RNA-Polymerase transkribiert, die der von Prokaryonten ähnelt (siehe auch Endosymbiontentheorie). Diese RNA-Polymerase wird auch im Plastidengenom kodiert, während eine weitere aus dem Cytoplasma importiert werden muss. In den Mitochondrien gibt es zwei RNA-Polymerasen, die beide importiert werden müssen. Sie sind mit dem kernkodierten plastidären Enzym verwandt.
In Archaea existiert für die Transkription wie bei den Prokaryonten nur eine RNA-Polymerase. Im Vergleich zu Prokaryonten ist diese aber komplexer. Größe, Anzahl der Untereinheiten und deren Aminosäuresequenzen sind eher mit den eukaryotischen RNA-Polymerasen vergleichbar.
Reifung der tRNA-Vorläufer
Die durch die Transkription entstehenden pre-tRNAs können Introns beinhalten. In Bacteria findet das Spleißen dieser Introns autokatalytisch statt, wogegen die Introns in Eukaryonten und Archaeen durch tRNA Splicing-Endonuclease entfernt werden.[8]
Bei Eukaryonten findet sowohl die tRNA-Synthese als auch ihr posttranskriptionales Processing (Spleißen, Methylierungen etc.) im Zellkern statt und die CCA-Sequenz 3’-Ende wird erst posttransskriptional dort angehängt.
Literatur
- Peter Karlson, Detlef Doenecke, Jan Koolman, Georg Fuchs und Wolfgang Gerok: Karlsons Biochemie und Pathobiochemie. 15. Edition (Auflage). Georg Thieme Verlag, 2005. ISBN 3133578154, 9783133578158. U. a.: S. 142 (803 Seiten).
Einzelnachweise
- ↑ Kim SH, Suddath FL, Quigley GJ et al.: Three-dimensional tertiary structure of yeast phenylalanine transfer RNA. In: Science (journal). 185, Nr. 149, August 1974, S. 435–40. PMID 4601792.
- ↑ Robertus JD, Ladner JE, Finch JT et al.: Structure of yeast phenylalanine tRNA at 3 A resolution. In: Nature. 250, Nr. 467, August 1974, S. 546–551. PMID 4602655.
- ↑ Peter Karlson, Detlef Doenecke, Jan Koolman, Georg Fuchs und Wolfgang Gerok: Karlsons Biochemie und Pathobiochemie. S. 142.
- ↑ WormBase: Release Notes – WS187 (25. Januar 2008).
- ↑ Spieth, J; Lawson, D: Overview of gene structure.. (Free full text) In: WormBook: the online review of C. Elegans biology. Jan 2006, S. 1–10. doi:10.1895/wormbook.1.65.1. PMID 18023127.
- ↑ Hartwell LH, Hood L, Goldberg ML, Reynolds AE, Silver LM, Veres RC.: Genetics: From Genes to Genomes. 2. Auflage, 2004. McGraw-Hill: New York, NY. S. 264.
- ↑ White RJ: Regulation of RNA polymerases I and III by the retinoblastoma protein: a mechanism for growth control?. In: Trends in Biochemical Sciences. 22, Nr. 3, 1997, S. 77–80. doi:10.1016/S0968-0004(96)10067-0.
- ↑ Abelson J, Trotta CR, Li H: tRNA Splicing. In: J Biol Chem. 273, Nr. 21, 1998, S. 12685–12688. doi:10.1074/jbc.273.21.12685. PMID 9582290.
Wikimedia Foundation.