- Megavirus chilensis
-
Megavirus chilensis Systematik Reich: Viren Familie: Megaviridae Gattung: Megavirus Art: Megavirus chilensis Taxonomische Merkmale Genom: 1,26 Mb ds-DNA Symmetrie: unvollkommen ikosaedrisch Hülle: vorhanden Wissenschaftlicher Name Megavirus chilensis (engl.) Taxon-Kurzbezeichnung MGVC Links Megavirus chilensis (MGVC) ist ein DNA-Virus, das 2010 von französischen Wissenschaftlern vor der Küste von Chile entdeckt wurde.[1] Es wurde der neu geschaffenen, vorläufigen Gattung der Megaviren zugeordnet[2] und ist deren einziger bekannter Vertreter. Das Virus ist ein entfernter Verwandter der Mimiviren (Acanthamoeba polyphaga Mimivirus = APMV)[3] und infiziert vermutlich Acanthamöben. Mit einem Genom von ca. 1,26 Mb besitzt es das bislang größte bekannte virale Genom. Es erreicht mit einem Durchmesser von ca. 700 nm fast die Größe eines kleinen Bakteriums und ist damit auch das morphologisch größte beschriebene Virus.
Aus der Analyse des Virusgenoms des MGVC ergaben sich Hinweise darauf, dass Mimiviren und Megaviren von einem gemeinsamen Vorfahren abstammen könnten, der durch reduktive Evolution aus Bestandteilen eines zellulären Genoms entstanden sei.
Inhaltsverzeichnis
Entdeckung
Megavirus chilensis wurde aus Meerwasser isoliert, das im April 2010 vor der Küste von Chile in der Nähe von Las Cruces von Jean-Michel Claverie und Chantal Abergel von der Universität Aix-Marseille gesammelt wurde. Wissenschaftler aus diesem Labor waren ebenfalls an der Charakterisierung des Mimivirus beteiligt. Das Virus wurde bei der Kokultivierung einer Reihe von Acanthamöben-Stämmen isoliert. (A. polyphaga, A. castellanii, A. griffini) Dabei wurde ein Verfahren benutzt, das von Timothy Rowbotham für die Isolierung intrazellulärer parasitischer Bakterien entwickelt worden war.[4] Der natürliche Wirt des MGVC ist vermutlich ein phagozytisches Protozoon, das im Meer- oder Brackwasser lebt.
Klassifikation
Das Virus ist bislang von der International Committee on Taxonomy of Viruses noch nicht klassifiziert worden. Vermutlich wird es als Mitglied einer neu zu schaffenden Familie Megaviridae beschrieben werden. Zu den Mitgliedern dieser vorläufigen Familie werden vermutlich solche Viren gehören, die mit dem Mimivirus und dem MGVC eine hinreichende Struktur- und Sequenzähnlichkeit besitzen. Das MGVC und das Mimivirus haben 594 orthologe Gene gemeinsam. Die meisten dieser übereinstimmenden Gene befinden sich in der Mitte der viralen Genome, jeweils flankiert von speziesspezifischen Abschnitten im 5′- und 3′-Bereich. Bezüglich der Aminosäurensequenzen sind die durch diese orthologen Gene kodierten Proteine zu etwa 50 % identisch. Megavirus chilensis gehört zu einer nicht-taxonomischen Gruppe von großen cytoplasmatischen Viren, bekannt als Nucleocytoplasmic large DNA viruses (NCLDV). Als Klassifiktionsmerkmal dieser Gruppe gilt die Tatsache, dass sie sich vollständig im Cytoplasma des Wirtes mithilfe sogenannter large virion factories vermehren, ohne die doppelsträngige DNA (wie sonst bei ds-DNA-Viren üblich) mithilfe der wirtseigenen DNA-Polymerasen im Zellkern der Wirtszelle zu replizieren.
Struktur
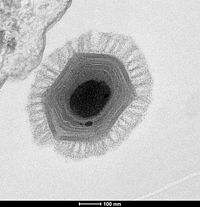
Das MGVC besteht aus einem Protein-Kapsid mit einem Durchmesser von 440 nm, das auf elektronenmikroskopischen Aufnahmen gesehen werden kann und ist von einem mehrlagigen Tegument-Material von 75 bis 100 nm Schichtdicke umgeben. Das Kapsid erscheint hexagonal, allerdings ist seine ikosaedrische Symmetrie nicht perfekt, da es mindestens ein sogenanntes „stargate“ enthält. Als „stargate“ wird eine Struktur bezeichnet, die einem fünfzackigen Stern ähnelt und die eine Öffnung bildet, durch die das Kernmaterial des Virus in das Cytoplasma des Wirts gelangt. Das Kapsid ist von zwei Membranen umgeben, die unterschiedliche virale Proteine enthalten.
Genom
Das Genom von Megavirus chilensis besteht aus einer linearen Doppelstrang-DNA mit einer Länge von 1.259.197 Basenpaaren. Es ist damit das größte bislang bekannte Virusgenom. Es ist um 67,5 kb größer als das Genom des Mamavirus und enthält vermutlich 1120 codierende Regionen, was mehr Proteinen entspricht als bei vielen Bakterien.
Das MGVC besitzt sieben Aminoacyl-tRNA-Synthetasen, Enzyme, die üblicherweise nur in zellulären Organismen vorkommen. Vier dieser Proteine sind auch aus dem Mimivirus und dem Mamavirus bekannt (diejenigen für die Aminosäuren Tyrosin, Arginin, Cystein und Methionin). Megavirus enthält zusätzlich die Aminoacyl-tRNA-Synthetasen für Tryptophan, Asparagin und Isoleucin. Das Cafeteria-roenbergensis-Virus enthält nur die Aminoacyl-tRNA-Synthetase für Isoleucin. Das MGVC enthält auch das Gen für eine besondere Variante des mismatch repair Enzyms MutS, das sonst nur in den Mitochondrien von Octocorallia vorkommt. Diese MutS-Version scheint nur bei Mitgliedern der Megaviridae-Familie vorzukommen.[5] Megavirus chilensis enthält – ähnlich wie das Mimivirus – Gene für den Stoffwechsel von Zuckern, Fetten und Aminosäuren.[6]
Replikation
Die Replikationsstadien des MGVC ähneln denjenigen des Mimivirus. Kurz nach der Phagozytose und der Freisetzung des inneren Kapsids in das Cytoplasma der Wirtszelle beginnt die Eklipse-Phase. Man findet dabei sog. cytoplasmatische „Seeds“, die in ihrer Größe dem Megavirus-Kern entsprechen. Diese Seeds entwickeln sich innerhalb von 14 Stunden zu Virion-Fabriken. Bis zur Lyse der Amöbenzelle und der Freisetzung der Viren dauert es beim MGVC gewöhnlich ca. 17 Stunden (im Gegensatz zu ca. 12 Stunden beim Mimivirus). Es werden dabei ca. 500 neue Viren pro infizierter Zelle produziert (im Gegensatz zu 1000 Partikeln bei Mimivirus).
Bedeutung der Entdeckung
Zwei Eigenschaften des MGVC sind von Bedeutung: Obwohl es nur 6 % größer ist als das Mimivirus, kann man doch annehmen, dass es nicht das größte Virus ist. Die Tatsache, dass drei weitere Aminoacyl-tRNA-Synthetasen in Virengenomen gefunden wurden, lässt vermuten, dass diese Enzyme nicht durch einen lateralen Gentransfer von anderen Viren erworben wurden. Die Analyse ihrer Sequenz zeigt eine Verwandtschaft mit eukaryotischen Genen an. Daher wird vermutet, dass das Genom dieser Viren ähnlich wie bei anderen Parasiten durch Genomreduktion aus dem Genom eines zellulären Organismus entstanden sein könnte. Man vermutet weiterhin, dass Megaviren evolutionär sehr alt seien, eventuell älter als die heutigen Eukaryonten, und gleichzeitig mit dem Zellkern entstanden sein könnten. Eine weitere Vermutung ist, dass Megaviren von einer zwischenzeitlich ausgestorbenen zellulären Domäne abstammen.[7]
Genomvergleich mit verwandten Viren
Virus Genom-Größe (bp) Gene Kapsid-Durchmesser (nm) Hülle Genbank-Nr. Megavirus chilensis[2] 1.259.197 1120 Proteine (abgeleitet) 440 ja (75 nm) JN258408 Mamavirus[8] 1.191.693 1023 Proteine (abgeleitet) 390 ja (120 nm) JF801956 Mimivirus[3][9] 1.181.549 979 Proteine (39 nicht-codierend) 390 ja (120 nm) NC_014649 M4[10] (Mimivirus « bald » variant) 981.813 756 Proteine (abgeleitet) 390 nein JN036606 Cafeteria-roenbergensis-Virus[11] 617.453 (730 kbp) 544 Proteine (abgeleitet) 300 nein NC_014637 Gemeinsame Merkmale großer Viren
Virus Aminoacyl-tRNA-Synthetase Octocorallia-ähnliche MutS Stargate[12] Bekannter Virophage[13] Cytoplasmische
Virion-FabrikWirt Megavirus chilensis 7 (Tyr, Arg, Met, Cys, Trp, Asn, Ile) ja ja nein ja Acanthamöben (Unikonta, Amoebozoa) Mamavirus 4 (Tyr, Arg, Met, Cys) ja ja ja ja Acanthamöben (Unikonta, Amoebozoa) Mimivirus 4 (Tyr, Arg, Met, Cys) ja ja ja ja Acanthamöben (Unikonta, Amoebozoa) M4 (Mimivirus « bald » variant) 2 (Met, Cys) nein ja resistent ja Acanthamöben (Unikonta, Amoebozoa) Cafeteria-roenbergensis-Virus 1 (Ile) ja nein ja ja Phagotrophische Protozoen (Heterokonta, Stramenopiles) Einzelnachweise
- ↑ http://www.pnas.org/content/early/2011/10/04/1110889108.abstract
- ↑ a b : Distant Mimivirus relative with a larger genome highlights the fundamental features of Megaviridae. In: Proceedings of the National Academy of Sciences. 2011. doi:10.1073/pnas.1110889108.
- ↑ a b : The 1.2-Megabase Genome Sequence of Mimivirus. In: Science. 306, Nr. 5700, 2004, S. 1344–50. doi:10.1126/science.1101485. PMID 15486256.
- ↑ : Isolation of Legionella pneumophila from clinical specimens via amoebae, and the interaction of those and other isolates with amoebae. In: Journal of Clinical Pathology. 36, Nr. 9, 1983, S. 978–86. doi:10.1136/jcp.36.9.978. PMID 6350372. Volltext bei PMC: 498455.
- ↑ : Two new subfamilies of DNA mismatch repair proteins (MutS) specifically abundant in the marine environment. In: The ISME Journal. 5, Nr. 7, 2011, S. 1143–51. doi:10.1038/ismej.2010.210. PMID 21248859. Volltext bei PMC: 3146287.
- ↑ : Mimivirus: The emerging paradox of quasi-autonomous viruses. In: Trends in Genetics. 26, Nr. 10, 2010, S. 431–7. doi:10.1016/j.tig.2010.07.003. PMID 20696492.
- ↑ : The challenge of microbial diversity: Out on a limb. In: Nature. 476, Nr. 7358, 2011, S. 20–1. doi:10.1038/476020a. PMID 21814255.
- ↑ : Viruses with More Than 1,000 Genes: Mamavirus, a New Acanthamoeba polyphaga mimivirus Strain, and Reannotation of Mimivirus Genes. In: Genome Biology and Evolution. 3, 2011, S. 737–42. doi:10.1093/gbe/evr048. PMID 21705471. Volltext bei PMC: 3163472.
- ↑ : Breaking the 1000-gene barrier for Mimivirus using ultra-deep genome and transcriptome sequencing. In: Virology Journal. 8, 2011, S. 99. doi:10.1186/1743-422X-8-99. PMID 21375749. Volltext bei PMC: 3058096.
- ↑ : Mimivirus shows dramatic genome reduction after intraamoebal culture. In: Proceedings of the National Academy of Sciences. 108, Nr. 25, 2011, S. 10296–301. doi:10.1073/pnas.1101118108. Volltext bei PMC: 3121840.
- ↑ : Giant virus with a remarkable complement of genes infects marine zooplankton. In: Proceedings of the National Academy of Sciences. 107, Nr. 45, 2010, S. 19508–13. doi:10.1073/pnas.1007615107. Volltext bei PMC: 2984142.
- ↑ : Distinct DNA Exit and Packaging Portals in the Virus Acanthamoeba polyphaga mimivirus. In: PLoS Biology. 6, Nr. 5, 2008, S. e114. doi:10.1371/journal.pbio.0060114. PMID 18479185. Volltext bei PMC: 2430901.
- ↑ : A Virophage at the Origin of Large DNA Transposons. In: Science. 332, Nr. 6026, 2011, S. 231–4. doi:10.1126/science.1199412. PMID 21385722.
Weblinks und weiterführende Literatur
- GiantVirus.org – an information resource on the genome of giant viruses.
- International Committee on Taxonomy of Viruses (ICTV) picture gallery - images of mimivirus
- : Giant Viruses. In: American Scientist. 99, Nr. 4, 2011, S. 304. doi:10.1511/2011.91.304.
- : Mimivirus relatives in the Sargasso sea. In: Virology Journal. 2, 2005, S. 62. doi:10.1186/1743-422X-2-62. PMID 16105173. Volltext bei PMC: 1215527.
- : Taxonomic distribution of large DNA viruses in the sea. In: Genome Biology. 9, Nr. 7, 2008, S. R106. doi:10.1186/gb-2008-9-7-r106. PMID 18598358. Volltext bei PMC: 2530865.
Kategorie:- Viren, Viroide und Prionen
Wikimedia Foundation.