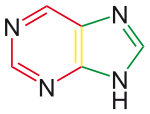
- Purinbasen
-
Strukturformel 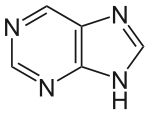
Allgemeines Name Purin Andere Namen - 3,5,7-Triazaindol
- 7H-Imidazo(4,5-d)pyrimidin
- Kurzzeichen R (Purin)
Summenformel C5H4N4 CAS-Nummer 120-73-0 PubChem 1044 Kurzbeschreibung Farblose Nadeln [1] Eigenschaften Molare Masse 120,11 g·mol−1 Aggregatzustand fest
Schmelzpunkt Löslichkeit löslich in Wasser und in heißem Alkohol, unlöslich in Ether, Chloroform, bildet Salze mit Säuren und Basen [1]
Sicherheitshinweise Gefahrstoffkennzeichnung [2] keine Gefahrensymbole R- und S-Sätze R: keine R-Sätze S: 22-24/25 LD50 WGK 3 - stark wassergefährdend [2] Soweit möglich und gebräuchlich, werden SI-Einheiten verwendet. Wenn nicht anders vermerkt, gelten die angegebenen Daten bei Standardbedingungen. Purin (R) ist eine heterobicyclische aromatische organische Verbindung mit vier Stickstoffatomen. Purine sind neben Pyrimidinen wichtige Bausteine der Nukleinsäuren. Sie sind nicht essentiell, sondern werden vom menschlichen Körper selbst gebildet. Tierische Lebensmittel enthalten viele Purine, da diese in Haut und Innereien in hoher Konzentration enthalten sind. Beim Menschen werden sie zu Harnsäure abgebaut und über die Nieren ausgeschieden; bei verschiedenen anderen Tierarten erfolgt ein weitergehender Abbau (beispielsweise zu Allantoin bei Rindern). Daher leitet sich auch der Name von lat. purus = rein und acidum uricum = Harnsäure ab, da es das „reine“ Grundgerüst der Harnsäure ist, die 1898 von Emil Fischer erstmals synthetisiert wurde.
Inhaltsverzeichnis
Struktur
Das Molekül kann als kondensiertes Ringsystem, zusammengesetzt aus den beiden Heterocyclen Pyrimidin (rot) und Imidazol (grün), aufgefasst werden. Deshalb lautet sein systematischer Name 7H-Imidazol[4,5-d]pyrimidin.
Das 7H-Purin steht mit seinem Isomer, dem 9H-Purin, in einem tautomeren Gleichgewicht:
Abgeleitete Moleküle
Purine
Übersicht
Werden die Wasserstoffatome in den Positionen 2, 6 und 8 durch andere Reste ersetzt, ergeben sich verschiedene substituierte Purine:
Purinderivate Name Grundstruktur R6 R2 R8 Purin 
−H −H −H Adenin −NH2 −H −H Guanin =O −NH2 −H Harnsäure −OH −OH −OH Hypoxanthin =O −H −H Xanthin −OH −OH −H 6-Purinthiol −SH −H −H 6-Thioguanin −SH −NH2 −H Xanthin =O =O −H Tautomerie
Auf Grund der Molekül-Abschnitte mit dem Grundmuster N=C–X–H (mit X = O, S oder NH) besteht die Möglichkeit der Tautomerie. (Siehe dort Lactam-Lactim-, Thiolactam-Thiolactim- und Ketimin-Enamin-Tautomerie):
Purin-Alkaloide
Vom Grundgerüst des Xanthins leiten sich auch einige Alkaloide ab:
Purinalkaloide Name Grundstruktur R1 R2 R3 Koffein 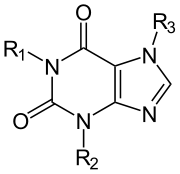
−CH3 −CH3 −CH3 Theobromin −H −CH3 −CH3 Theophyllin −CH3 −CH3 −H Rezeptoren
Purine binden an spezifische Rezeptoren in der Zellmembran, sog. purinerge Rezeptoren. Es gibt ionotrope und metabotrope purinerge Rezeptoren. Der physiologische Agonist dieser Rezeptoren ist ATP.
Biologische Bedeutung
- Die Harnsäure ist das Endprodukt des Purinstoffwechsels.
- Sind Adenin und Guanin in Position 9 mit dem C-1 Atom der Ribose (bei der DNA mit Desoxyribose) verknüpft, ergeben sich die Nukleoside Adenosin und Guanosin. Durch Veresterung der Ribose mit Phosphat entstehen die Nukleotide, die Bausteine zahlreicher physiologisch wichtiger Moleküle sind. (Siehe dazu auch die B-Vitamine: Nicotinsäure, Riboflavin und Pantothensäure sowie AMP, ADP, ATP, RNA, DNA, cAMP, NADPH, NADH, FAD, Coenzym A, Succinyl-Coenzym A.)
- Durch Salpetrige Säure (HNO2) wird die Amino-Gruppe in eine Hydroxylgruppe umgewandelt. Dadurch entsteht aus Adenin Hypoxanthin und aus Guanin Xanthin. Wirkt Salpetrige Säure auf die DNA (als Mutagen) ein, führt diese Änderung dazu, dass es bei der Vermehrung der DNA (Reduplikation) zu fehlerhaften Basenpaarungen und damit zu einer veränderten Basensequenz kommt, die zu veränderten Proteinen und damit zu einem veränderten Phänotyp führen kann.
- In der tRNA finden sich auch ungewöhnliche Purinbasen (Langbasen): A+ entsteht durch Anlagerung eines Protons an Position 7, dadurch wird das Stickstoffatom an dieser Stelle positiv geladen. Die Stickstoffe des Ringsystems können methyliert sein, wodurch sie ebenfalls positiv geladen sind. Beispiele: m1A, m7A, m7G. Bei m2’G ist das C-2 Atom der Ribose methyliert. Die entsprechenden Basen werden erst nach der Transkription modifiziert und beeinflussen die Genauigkeit der Translation sowie die Aktivität und Stabilität der tRNA. (siehe auch: Nukleoside)
- trans-Zeatin (N6-(Δ2’-Isopentenyl)-aminopurin) ist ein natürlich vorkommendes Cytokinin.
Biosynthese
De novo-Biosynthese
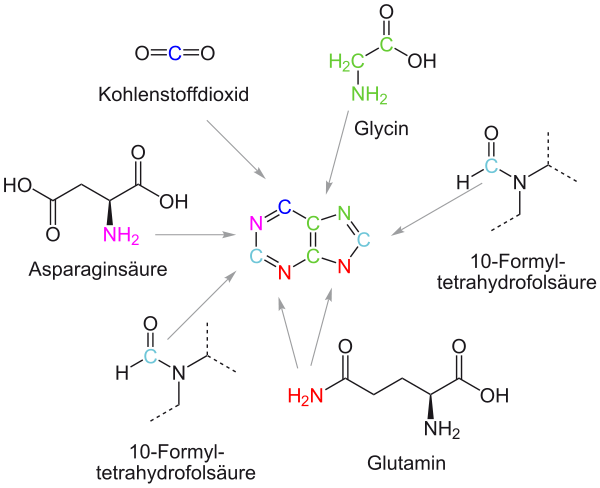
Die Purine werden im Organismus nicht als freie Moleküle synthetisiert, sondern stets als Nukleotide. Ausgangsmolekül ist das α-D-Ribose-5-phosphat, ein Zwischenprodukt des Pentosephosphatzyklus. Darauf wird das Grundgerüst des Purins schrittweise aufgebaut, wobei verschiedene Moleküle die einzelnen Bestandteile liefern:
Das Endprodukt dieser Synthesekette ist das Inosinmonophosphat (IMP), das Nukleotid des Hypoxanthins, welches in weiteren Schritten zu den Nukleotiden des Xanthinosins, Adenosins oder Guanosins umgebaut wird.
Wiederverwertung der Basen (Salvage-Pathway)
- Hauptartikel: Salvage-Pathway
Beim Abbau der RNA entstehen neben den Mononukleotiden auch freie Basen und Nukleoside. Die Mononukleotide können dadurch wieder zurückgewonnen werden, dass die Purinbasen mit phosphorylierter Ribose und die Nucleoside durch Kinasen ihre Phosphatgruppe wieder erhalten.
Im Verbund mit Ribose / Desoxy-Ribose: Reduktion der Ribonukleotide
Um als Bausteine der DNA dienen zu können, muss die Ribose (Zucker) der Nukleotide am C-2 Atom reduziert werden, die Hydroxyl-Gruppe wird durch ein Wasserstoffatom ersetzt (nun heißt die Ribose Desoxy-Ribose) und es entstehen die Desoxy-Ribonukleotide. Bei RNA hingegen wird Ribose und nicht Desoxyribose als Zucker verwendet. RNA kommt gewöhnlich nicht als doppelsträngiges Molekül (wie DNA) vor.
Abbau der Basen
Von den Nucleosiden und Nucleotiden werden als erstes die Basen abgespalten. Diese werden zu Harnsäure oxidiert. Diese ist bei Landreptilien, Vögeln, vielen Insekten und Primaten das Endprodukt, das über den Harn ausgeschieden wird. Andere Tiere bilden aus der Harnsäure Allantoin, Harnstoff oder Ammoniak.
Medizinische Bedeutung
Krankheiten
Je nach Ort der Störung im Purin-Stoffwechsel ergeben sich verschiedene Krankheitsbilder:
- Das Lesch-Nyhan-Syndrom ist auf einen Mangel eines Enzyms (Hypoxanthin-Guanin-Phosphoribosyl-Transferase, HGPRT) innerhalb der Wiederverwertung der Basen (Salvage-Pathway) zurückzuführen. In dessen Folge häufen sich die Substrate 5-P-Ribosyl-PP sowie die Purine Hypoxanthin u. Guanin an, wobei letztere damit auch zu einer Anhäufung von Harnsäure führen. Diese verursacht Gicht, geistige Behinderung und Verhaltensauffälligkeiten.
- Die Gicht ist eine Folge von Hyperurikämie, eine erhöhte Konzentration von Harnsäure im Blut (mehr als 0,4 mmol/l). Unterhalb dieser Konzentration stehen im Blut genügend Eiweiße zur Verfügung, um die in Wasser schwerlösliche Harnsäure zu transportieren und ihre Ausfällung zu verhindern. Ist der Harnsäure-Wert zu hoch, reicht dieses Schutz-System nicht mehr aus und es kommt zu Ablagerungen in Gelenken, Sehnenscheiden und Nierenmark.
- SCID (schwerer kombinierter Immundefekt) ist auf eine 50-fache Erhöhung der dATP-Konzentration zurückzuführen. Dadurch ist die empfindliche Balance in der Konzentration der DNA-Bausteine gestört und es kommt zu Störungen der DNA-Synthese, die vor allem die Zellen des Immunsystems (T- und B-Zellen) betrifft.
Arzneistoffe
Purin-Derivate und Purin-Analoga spielen als Antimetaboliten eine Rolle: Azathioprin unterdrückt das Immunsystem, 8-Azaguanin, 6-Purinthiol und 6-Thioguanin werden gegen bestimmte Krebsformen eingesetzt, Allopurinol gegen Gicht. N-Hydroxy-Purin und Purin-N-Oxide wirken carcinogen (krebserregend).
Einzelnachweise
- ↑ a b c Thieme Chemistry (Hrsg.): Römpp Online. Version 3.1. Georg Thieme Verlag, Stuttgart 2007.
- ↑ a b Angaben des Hersteller Sigma-Aldrich, 27.12.2007
- ↑ Purin bei ChemIDplus
Siehe auch
Weblinks
- MDR.de - Infos + Puringehalt einiger Lebensmittel
- MedizInfo.de Lebensmitteltabellen für Purine und Harnsäure
- PurinTabelle.de Lebensmitteltabellen für Purine und Harnsäure
- Müller, C.: Gichterkrankung - Purinarme Kost gegen Schmerzen. aus: UGB-Forum 2/00, S. 68-71, UGB-Verband, 2000
Wikimedia Foundation.